探索・設計・発現をつなぐ基盤技術開発と人材育成の現場:CTO中村の視点より(社員インタビュー)
はじめに
本記事は、弊社noteへ2024年9月に掲載されたインタビュー記事を、より多くの方にご覧いただけるよう、当社公式テックブログにも転載したものです。内容は掲載当時のものとなります。
本文
ーー記念すべきインタビュー第1回目です。中村さん、よろしくお願いいたします。
早速ですが入社の経緯を教えてください。・・・といっても、中村さんは創業メンバーですよね。
そうですね。東京工業大学在学中に株式会社digzymeを、渡来さん(注1:渡来直生さん、digzyme代表取締役CEO)、山田先生(注2:東京工業大学准教授山田 拓司先生、digzymeの取締役CSO)と共同創業しました。
創業のキッカケとしましては、digzymeの創業の元となった研究を、山田研(注3:東京工業大学生命理工学院 山田研究室)で実施していて、ビジネスの可能性を感じたことです。
ーー創業の元となった研究・・・是非、もう少し詳しく教えてください。また、どんなところに
ビジネスの可能性を感じたのでしょうか?
現在のdigzyme Moonlight™の元にあたる研究で、『酵素を探索します』という内容でした。
長瀬産業さんと山田先生が共同で研究をしていたところに、僕も入って追加で解析をしました。
具体的には長瀬産業さんから
「こういう酵素が欲しい」
というオーダーをいただき、それをデータベースから探す・・・ということをやっていて。それで順調に酵素がみつかっていたので、まず論文にしましょうということになりました。
その後、もしこの探索技術を『他の部分』に適用したらどうなるのか?という考察を深めていったんです。
ーー他の部分とは?
この世界でまだ触媒する酵素が見つかっていない化合物の合成反応をみつけられるのか?というところですね。これが長瀬産業さんとの研究で成果が上がりまして。ある植物が持っている『特殊な化合物を作るための酵素』についての研究だったのですが。毎回その酵素を、植物をすりつぶして抽出するのは大変じゃないですか。
なので、微生物の酵素反応を使って、酵素化学的にできたら簡単なのでは?ということで、それが可能な酵素を探索してみたんです。そしたら、結構たくさん見つかったんですね。
それも、まだ発見されていない、世の中では売られていないような化合物を合成するような酵素が、結構たくさん。
ーーすごいことですね。
はい。それからは、作るのがすごく難しい化合物をこれらの酵素を使えば、実は簡単に作れるんじゃないか?ということを論文のなかで議論して。
山田先生に報告したところ、こういった酵素が欲しい人々は世の中にかなりいらっしゃるんじゃないか?という話になり、ビジネスとしてやったら面白いのでは、と。
さらにタイミングがすごく良かったのですが、その当時、ちょうど渡来さんが、ベンチャー企業の立ち上げに興味をもって活動していて。
そのような流れもあり、山田先生と、渡来さんと、僕とで活動をスタートしたら早速、『そういう酵素を探す技術があったら使いたいです』というところから、複数お声がけ頂いたんです。なので、これだけニーズがあるならば創業した方がいいだろう、ということで創業に至りました。
ーー確かに、従来の酵素開発では、目的に合った酵素の遺伝子を見つけるまでに、膨大なトライアンドエラーが必要でした。
そのため、偶然の発見に頼らざるを得ない不確実性と、莫大な開発コストが問題となっていましたが・・・この点を考えると、かなりのニーズがありますよね。
創業後はどのような業務に携わってこられたのですか?
創業直後は、コアテクノロジーにあたる酵素探索ソフトウェアの開発作業を行いつつ、次はそれをさらに応用して、より会社の強みになる部分をどう開発していくか?というところを進めていきました。
すでに研究開発のテーマもいくつか自社内であがっていたので、それらの開発を含めて。
でも僕は、創業から半年間はまだ学生だったのですが、卒業後は2年近く製薬会社に勤めていたんですね。
なのでその期間は兼業しながら技術開発のディスカッションなどを中心に参加していました。
ーーその後、digzymeにフルコミットする形になられたんですよね。
はい。digzyme1本になったあたりで、ちょうどNEDO(注4:国立研究開発法人 新エネルギー・産業技術総合開発機構)のプロジェクトが始まりました。
そこでspotlight(注5:酵素機能改良プラットフォームdigzyme Spotlight™)のデザイン・・・『こういうふうな機械学習で、こういうモデルをつくったら上手くいくでしょう』という部分を渡来さんたちと一緒に考えて、予算が取れたものを実際に研究員たちに配分して、誰々がどこを作ってくださいね、という感じで開発を進めていきました。
ーー自然と各プロジェクトのリーダーを担うようになっていったそうですね。CTOとしての、採用や教育も。
はい。メンバーが少しずつ増えていく過程で、新入社員へのDRY研究技術の教育も行なっていくようになりましたね。一番最初は、創業直後にアルバイトで入ってもらった礒崎(注6:Principal Investigator、礒崎達大さん)に・・・彼はもともとDRYではなくWET研究の出身だったので、僕と渡来さんでDRYの技術を教えました。
ーーそうだったんですね。DRY技術の教育って、具体的にはどのように進めるものなんですか?
基本的には実例を試すのが一番、だと思っています。プログラミングの勉強って一般的に例えば『1+1=2』みたいな計算をプログラムにやらせたらどうか?というようなことを勉強するんですが・・・
そうするとあんまり身にならないというか、やっぱり『それって現実にどう活かされるんですか?』
いう感じがしちゃうので。
ーーなるほど。
現実の課題を解いていかないとあんまり面白くなくて、身にならないことが多いんですよ。なので、現実的な課題をアサインすることが、教育的にもよいと思っています。
例えば、僕が作っている研修用の資料には、過去に僕自身があたった課題が書いてあります。それを解いて、一通りやってみてもらう、という手法で教育を進めていますね。
さらに最近だと、現実にお客様から頂いた課題をテストケースにして、高山さん(注7:Principal Investigator、高山裕生さん)や礒崎と一緒に解いてもらうやりかたが多いです。
ーー詳しく教えていただきありがとうございます。最近の業務はいかがですか?
個別プロジェクトのマネジメントや、研究リソース管理などが主な仕事になっています。
ーーマネジメントを行うのって、中村さんにとってはどんな感覚ですか?
僕の場合は、マネジメントももともと苦手な項目ではなく、、、学生時代にアルバイトしていたときも、バイトリーダーなどでメンバーの管理などをよくしていたので。そもそも、物事のコアな部分に関わっていないと、気が済まない感覚があるんですよね(笑)
製薬会社勤務のときも、例えば末端で実験・研究だけをしているというよりプロジェクトの詳しい話や本質的なところまで入っていかないと納得がいかず、満足しない方でしたので。
もちろん研究自体は好きなのでやっていたいなという感覚はあるんですが、何も知らずに手を動かすことができないタイプなので・・・
そういう意味でも自然とマネジメントをやっているのかなーという感じがしますね。
でも、これらも礒崎と高山さんに引き継ぎつつあるので、新規技術開発のリードとか、基盤技術の開発がまた僕のコアな業務に戻りつつある、というところですね。
もちろん、DRY・WETのメンバーと、新規技術としてはどういうものを持つべきか?
あるいは、digzymeとしての強みをさらに伸ばすためのアイデアを考えたり深めたりすることは、常々行なっています。
ーーdigzymeとしての強みが、さらに伸びていくためのアイデア、とても気になります。
そうですよね。今後より伸びていくためにという部分に関しては、事業部の皆さんを中心に考えていただいた『事業領域として拡大したい箇所』において必要なソフトウェアだったり、必要なWETの技術などを議論しながら作っていくこともしっかりやっていきたいですね。
それと僕自身は、各プロジェクトのなかで基盤として作ったものがうまく活用されてプロジェクトが進んでいくように動いています。
ーー中村さんは、digzymeで働いていて、どんなところにやりがいを感じますか?
新しい技術を現実の課題に適用しながら、足りない部分を開発したり、アップデートしていったりするところにやりがいを感じます。
例えば、digzyme Spotlight™の開発も、AI、機械学習を用いて酵素を改良しようという試みを実践している存在が世の中でほぼ皆無ななか、行っていました。
それまでも、一般的には酵素の立体構造をみて、『基質と相互作用するタンパク質の場所を変えたら、基質にも影響はあるだろうから変えて、活性をあげましょう』というような研究はされていたんですけれども。
ーー機械学習を用いてという手法は、一般的にとられていなかったわけですね。
はい。それでも、酵素の性能が足りないのでもっとよくしたいというオーダーは多くて、酵素業界全体の課題だったんです。
何かの課題に直面して解決していくという作業が僕は好きなので、
「digzymeだったら、活性を上げる変異体をAIで予測してデザインできそう」
という議論をしながらプログラムを作って、開発していく過程にとてもやりがいを感じました。
ーーSpotlightの開発ケースは相当やりがいに繋がったということですね。
そうですね。
でも、課題そのものは、小さい規模でも大きい規模でも楽しいんですよ。
Spotlightのケースはちょっと大きめですけど・・・日々出てくるちょっとした『これってめんどくさいよね』みたいなものを直して、上手くまわるようになっていく過程自体がとても好きです。
どんな規模でも、新規で技術開発して課題を解決していく『改良』というところにやりがいを感じます。
ーーなるほど。大小問わず課題を解決することにやりがいを感じる中村さんに、digzymeは支えられているんですね!せっかくなので、この流れでSpotlightの独自性についても触れてみたいのですが・・・
開発メンバーが、それぞれの経歴を存分に活かしたからこそ仕上げられたプラットフォームだと耳にしています。この辺りについて、詳しく教えていただけますか。
Spotlightは酵素について、機械学習のアルゴリズムで
「こういう風にやったら、変えるべき場所が予測できるだろう」
というプログラムです。僕は学生時代はもちろん、製薬会社のなかでも機械学習の研究をずっとやっていたので、その知識を活かして。
配列に詳しいメンバーとしては渡来さんや彦有さん(※注8:Informatics Specialist 鈴木彦有さん)。彼らはゲノムとして遺伝子、タンパク質配列を解析するということを研究室でずっとやっていたので。
あとは田村さん(注9:Informatics Specialist 田村 康一さん)ですね。彼は立体構造のデータにすごく詳しいんです。なので、配列と立体構造のデータに詳しい、渡来さん、彦有さん、田村さんの三人に、どういう特徴を学習させたらいいだろうということを考えてもらって、僕の方では『機械学習のモデルにはこういうやり方がありますよね』ということを考えて、最終的には礒崎に実装をしてもらって、仕上がったんです。
ーーまさに『叡知を結集』という感じで感慨深いです。たくさんお聞きできたので、次は仕事で苦労したことや、乗り越えられたキッカケについて伺っても良いですか?
苦労というか、採用活動は結構大変だなと感じながら行なってきました。
会社にとっても、また、採用される個人の人生にとっても、大きいことですしね。重たいことだな、という認識があります。
そんななか、digzymeの未来を担っていただく人材の採用に関して、どう判断したら・・・というところを、かなり悩みながらやってきました。
幾度かの採用活動を経て、最近はようやくコツを掴んだ感じがあります。
渡来さんは、面接時における質問の内容など、採用活動が上手な印象があるので・・・そこは真似させていただいて。
ーーちなみにどんなかたを採用することが多いですか?
やっぱり、話していて違和感がない人・・・これは絶対ですね。
こちらの質問に対して想像の範囲の回答はもちろん、そこを超えた範囲で応えてくださるかたは、前向きに採用したいなという気持ちになります。逆に、思っていたよりも二手三手後ろで止まっている回答をなさる場合は、ちょっと難しいかな、とは思っています。
また、トラブルシューティングが上手な方であることが望ましいです。特にWETの研究は、失敗がつきもの。
DRYはなにかうまくいかなかった時にすぐやり直せますし、僕自身がアイデアを出しやすい分野でもあるのですが、WETの実験はやり直そうと思った時に『また1週間失くなります』・・・など、大幅なスケジュール変更を余儀なくされるわけです。
さらに正直なところ、僕自身がWETにそこまで詳しいわけじゃないので、なにか上手くいかなかった時に『じゃあどうしたらいいと思いますか』と一緒に考えるフェーズにおいて、やっぱり僕以上に詳しくて、ご自身で考えて動けるかたがいらっしゃると嬉しいです。
実験的なトラブルが出ることは本当に多々あるので、しっかり対処できるかという・・・
過去に失敗したケースにどう対応したかなどを伺い、トラブルシューティングが上手な方を採用できるように努めています。
ーーなるほど。教えていただきありがとうございます。
WETのお話が出ましたが、そうしてメンバーを採用してきたからこそ、digzymeのWETの強みって、あれだけすごいんですね。
そうですね。
digzymeのWETに関しては『意外となんでもできる』のが強み、だと思っています。例えば『こういう酵素の評価をしたいです』となったときに、論文を読みながら開発をして、試して、実際に発現させて
評価をして・・・ということをやっていくわけなんですけれど、それって、もちろんちゃんと『研究ができる』方じゃないと難しくて。
例えば、僕が軽く論文読んで追試してくださいって言われて、『実験してみよう!』って軽々できることではないんですよね笑
そこを自然にこなせてるっていうのは、実はものすごくレベルの高いことなんです。
逆にいうと、うちがDRYで解析したものをWETの操作が理由でこれ以上進めません、ということはほぼないです。サラッと言っていますが、これも実はものすごいことです。
ただ、技術はとても高いのですが、リソース的に強いか・・・というところはそうでもなくて、いろんな企業さんとかアカデミアの先生がいらっしゃいますけど、そこと比べるとやっぱり、『特別な微生物株を持っています』とか『特別な遺伝子組み換えが技術があります』とかではなく、あくまで使っているのは公開されているものと同じものを使っているので。
リソース的な強さは正直ないですけど、人員的な研究員としての能力はとても強いと自負しています。
ーーなるほど。心強いですね。
はい。
ちなみにDRYって『因果関係はわからない』ものなんです。どっちが理由で、どっちが結果か?っていうことに関してわからない部分が多いので、僕らが酵素の解析をするときも偽陽性についても考慮しながら進めます。
そこですごく可能性の高いところまで絞っていくのですが、そのあとはWETの技術の高さにとても支えられています。
例えば大腸菌を使うにしても、1株だけじゃなくて何株も用意して、別の生物も色々用意して・・・など、潜り抜けて実験していく技術がとても優れているので、ありがたいです。

ーー今後は、どのようなことにチャレンジしてみたいですか?
基本的には、プロジェクトのステージが進んでいって、上市して何かの役に立ってくれれば、と思っています。
今開発している酵素が、製品になって表に出てくるのは楽しみです。『実はこの製品に入っているんだよ』と言えるくらいになったら嬉しいなと思っていますね。
技術的なチャレンジでいえば、プロジェクトのステージが進んでいったからこその課題が出てくるはずなので、解決していきたいです。
例えば、『こういう化合物を、こういう別の化合物に変化させたい』という時に、『酵素量はこれくらいの量で』『どれくらいの効率で』という数値が現状よりは具体的になってくるはずです。
それを達成できるorできない、が近い未来の重要課題になると予想しています。
さらに次のステージでは“大量生産“が待っています。『製品の価格がいくらなので、そのコストで作るためには、培養液はこれくらいの量で・・・』『この量の酵素が作れないといけない』などの生産性能の目標が見えてくるはずです。
これらの達成は事業継続にも関わる部分ですので、しっかり解決していく予定です。
ーー目標値がもう少し厳しく明確化されていくと、生産性能向上や安定が肝になってくるということですね。他にもなにかチャレンジしたいことはありますか?
完全に人工の酵素の開発はしてみたいですね。
ーー完全に人工の酵素?
『完全に』っていうのは、また難しいですけれども(笑)
普通は天然に存在する微生物が持っている酵素の情報をベースに作るとか、それを改良するというやり方ですが、データからいきなり酵素をデザインするということが、今はAI技術を上手く使うと、少しできるようになってきているんです。
そうすると、もしかしたら何かの微生物の酵素には、ちょっとだけ似ているかもしれないですけど、『ベースの酵素』があるわけではなく、パソコン上でポチポチ作業をしていたら酵素がデザインされて、
それがどの微生物由来でもないもの、というのが作れます。
ーーなるほど。
まぁ、これを例えば食品に入れて、実際に食べたいか?と言われると難しいかもしれないですけども(笑)
今は天然にあるものがベースなのでそこに縛られていますが、それを完全に脱却してオリジナルな酵素を作ることができるので。
実際に何かに使えるかどうかは置いておいても、新しい感じがして面白いですよね。
あとは無細胞系などの微生物に依存しない開発はしてみたいですね。
無細胞系は今社内でもちょっと使っていたり、議論にもなっていたりしますが、基本的にはWETの過程において微生物で遺伝子組み換えして発現・・・という流れが中心なので、やはり『タンパク質が発現しないです』ということが往々にしてあります。というところで無細胞系・・・ーもちろん無細胞でも発現しないことはあるのですがー比較的抑えられることもあるので、そういう意味でトライしてみたいです。 とにかくバイオ特有の不明瞭さがなくなると嬉しいな、と。
ーーバイオ特有の不明瞭さ、というと?
バイオの実験って『なんかよくわからないけど上手くいかない』ということがよくあるんですよね。この『よくわからないけど上手くいかない』が無くなるとすごく嬉しいよね、と思っています。
どうやったら確実にできます、という話では全然ないので、本当に夢の話をしていますって感じなんですけれど(笑)
例えば『培養の条件』一つとっても、この培養条件が一番良いです、ということが理論的にわかるわけじゃなくて、ある微生物を培養する時に、培地にどういう成分をどれくらい入れれば良いか、みたいなことは何度も何度も実験して、この組み合わせが最適っていうのを探し出すんですよね、やり方としては。理論的にこれだ!ってわかるわけじゃなく、なんでかよくわからない・・・。
培養していて、上手く増える時もあれば、そうでないときもある。タンパク質も、作ってみるとあんまり生産量も一定ではなく、多い時もあれば少ない時もある・・・という感じで結構ズレがあるんです、
バイオの実験って。
『失敗する』というのは、極端に『不明瞭なケース』ですけれど、上手くいっているときでも『特別上手くいっているとき』も『微妙だけど上手くいっているとき』もあり、誤差が大きい。
ーーなるほど、それは確かに不明瞭ですね・・・。
そんな不明瞭さのなかでもまず『宿主ごとに上手く発現しない』というのは、やはり大きな課題なので・・・
このあたりが改良できるように、『この世の酵素を、何でも発現できるシステムみたいなものが
あったらいいね』という意味で、あるとしたら無細胞なのかな、という気はしています。
ーー何でも発現。夢がありますね。詳しくありがとうございます。最後に、digzymeに応募を考えている未来の仲間に一言あれば、お願いします。
「バイオの曖昧な課題の解決に最先端技術とアイデアで一緒に挑戦しましょう!」
ーー中村さん、ありがとうございました。
終わりに
▼オリジナル記事はこちら(note)
https://note.com/digzyme/n/n4cb24197110b
「digzyme Custom Enzyme Lab」で期待される実用化例:糖鎖構造構築および難分解性物質へのアプローチ
はじめに
2025年5⽉21⽇(水)、22(木)、23 ⽇(金)
と3日間に渡り開催されたifia JAPAN 2025。
昨年と同様、弊社の代表取締役、渡来が出展者プレゼンテーションを行いました。
その様子をYOUTUBEで公開いたしましたので、ぜひご覧ください。
今回の出展者プレゼンテーションでは、2025年5月21日にローンチされた「digzyme Custom Enzyme Lab」についてご紹介しました。当日は、DRY技術(バイオインフォマティクス解析)とWET技術(実験検証)という二つの技術的アプローチに触れつつ、プラットフォーム全体の概要をお伝えしました。
本記事では、その中で取り上げた『「digzyme Custom Enzyme Lab」で期待される実用化例』2件について、渡来の視点を通じて、各事例における技術的ブレイクスルーやin silico設計の裏側をQ&A方式で詳しくご紹介します。
当日のプレゼンテーションでは全体像のご紹介にとどまりましたが、本記事を通じて、「digzyme Custom Enzyme Lab」の実力と可能性をより具体的にご理解いただける内容となっております。ぜひ最後までご覧ください。
まずは一つ目の事例についてです。

「digzyme Custom Enzyme Lab」で期待される実用化例1
Q.この成果の最も大きな意義は何だと考えていますか?
A.糖質は、構成する糖の結合様式の違いによって物性の差が生まれます。in silico技術で、目的の糖鎖構造を作る酵素を狙って探索ができた例は学術的にも稀で、かつ10個という少数の実験で発見できたことは非常に価値が高いと考えています。
Q.これまでのアプローチと比べて、今回のアプローチは何が革新的だったのでしょうか?
A.本件は、結果的には、当時のAlphaFold2に代表される深層学習(DL)ベースの構造予測技術に対して、弊社独自の詳細な分析技術を適用した点が役に立ちました。これまでのhomologyベースのモデルでは、糖鎖構造を作り分ける微妙なタンパク質構造の違いまでは予測することが困難でしたが、当時のAI技術によりそれらの特徴を一定程度捉えられたと考えています。
(なお、現在の生成モデルを用いた最新のAI技術とはギャップがあるため、本稿では便宜的に「AI」とまとめて表現しております。)
Q.チームや関係者のどのような努力がこの成果につながったと思いますか?
A.担当の研究員が、顧客ニーズを詳しく深堀りして当該酵素のスクリーニング基準をうまく設定し、基盤開発メンバーと連携することで解析プログラムを個別に作成し、この成果につながりました。当社では、既存プラットフォームだけで達成できない課題に対しても、フレキシブルにツール開発を行える点が強みだと思っています。
次に、三菱ケミカル株式会社と共同で行った、二つ目の事例についてです。
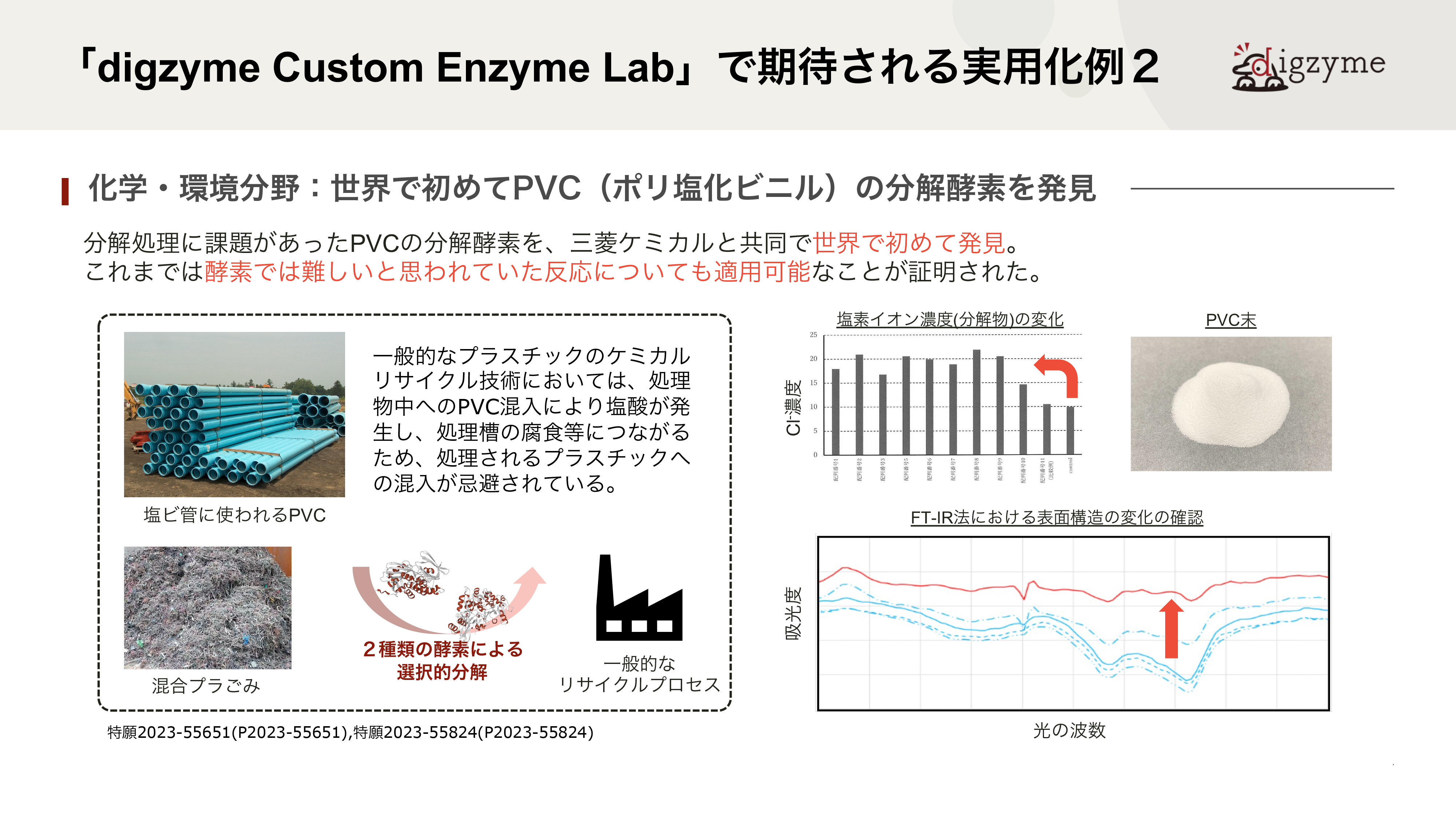
「digzyme Custom Enzyme Lab」で期待される実用化例2
Q.この成果の最も大きな意義は何だと考えていますか?
A.PVCは20世紀から本格的な生産が始まった自然界にない物質で、天然の微生物が進化の過程で分解機構を獲得していないと仮定すると、最適化された酵素は天然からは見つからないはずです。一方で、生物は休眠遺伝子を含めて”最適化されていない”さまざまな遺伝子をゲノムに保有しており、結果として環境変化への適応に活かされるとされています。本件は、その環境適応に寄与しうる酵素をin silicoで人工的に見つける問題とも捉えられるため、難易度が高いテーマでした。
Q.従来、このような酵素を発見するにはどれほどの時間やコストが必要でしたか?
A.近年、人工的なプラスチックの分解酵素を、集積培養に近い形で見出す研究がいくつか見られます。例えば、ある樹脂を海底に一定期間沈めておき、引き上げた後に分解の様子を観察したり、バイオフィルムに含まれる微生物を単離培養したりします。うまくいく場合には、分解酵素を持つ微生物が見つかるので、ゲノム解析やBACライブラリ作成などを通じて分解酵素を同定することが出来ますが、分解が遅いという性質上、どうしても年単位での時間がかかってしまいます。もちろん、分解が観察されず、うまくいかないケースも数多くあると思っています。in silicoでの探索は長くても半年程度で済むため、このような実験時間が長くかかってしまう対象に対してもある程度効果的に用いることが可能です。
終わりに
渡来は、今回の出展者プレゼンテーションを振り返りながら、次のように語っています。
「『digzyme Custom Enzyme Lab』でも、事前の準備期間の中で、これらの共同研究ケースのようなin silicoライブラリを作成して進めていくことができます。高精度のライブラリから精製酵素を試したいお客様におすすめしたいサービスです。」
このコメントが示すように、バイオインフォマティクスを基盤とした酵素設計アプローチは、限られたリソースの中でも実用的な酵素開発を加速しうる可能性を持っています。
今後さらに、酵素の多様な分野への応用が進む中で、「digzyme Custom Enzyme Lab」はその中核を担う技術基盤として重要な役割を果たすと考えられます。
ifia JAPAN 2025会場で頂戴したご質問への回答まとめ
はじめに
食品事業部の村瀬です。
弊社は、2025年5月21日(水)〜23日(金)に東京ビッグサイトで開催された「ifia JAPAN 2025 第30回 国際食品素材/添加物展・会議」(主催:食品化学新聞社)に、昨年に続いて出展いたしました。
会期中は、弊社の技術に高い関心をお持ちの多くの来場者の方々と、直接お話しできる貴重な機会となりました。
出展ブースでは、そうした皆さまに向けて、弊社の最新の取り組みをご紹介していましたが、なかでも大きな目玉となっていたのが、新しいソリューション「digzyme Custom Enzyme Lab」のローンチについてです。(詳しくはプレスリリースをご覧ください:https://prtimes.jp/main/html/rd/p/000000018.000050097.html)


ローンチにあたり、想定を上回る多くのご反響をいただき、出展ブースでは連日、多くの方から具体的なご質問やご相談をいただき、終始活発な対話の場となりました。
そこで今回のテックブログでは、「digzyme Custom Enzyme Lab」のローンチを記念した特別編として、会期中にいただいたご質問の中から、特に多かった内容をピックアップし、回答と合わせてQ&A形式でご紹介します。
本ソリューションにご興味をお持ちの方はもちろん、「酵素を使った開発に関心はあるけれど、何から始めたらよいのかわからない」という方にも、ヒントとなる内容です。ぜひ最後までご覧ください。
Q:「digzyme Custom Enzyme Lab」は、どんな開発テーマに利用できる?
A:「digzyme Custom Enzyme Lab」は、「現行の酵素を用いた製造法の効率向上」のような具体的な開発テーマから、「(酵素を用いた)新規食品素材開発」のような抽象度の高い開発テーマにもご利用いただけるソリューションとなっております。精製酵素サンプル提供とお客様での酵素評価のフィードバックを繰り返し行うことで、随時開発方針の調整が可能です。
Q:提供される精製酵素サンプルについて、どんな情報が提供される?
A:基本的な酵素活性の有無、プロファイル(至適温度、至適pH、熱安定性、pH安定性)の確認を弊社で行い、サンプル提供と併せて提出いたします。実際の評価系での実証実験については、お客様ご自身で確認することができます。
Q:提供される精製酵素のサンプル量はどれくらい?
A:開発テーマごとにもご相談となりますが、典型的には、酵素溶液として~mL、タンパク質として~mg単位でのご提供となります。
Q:初期開発期間はどのように決まる?
A:お客様がご希望するテーマについて、事前に弊社で開発方法の検討・調査を行い、初期開発期間を決定いたします。基本的には、2〜6ヶ月の間で初期開発(in silico酵素デザイン〜初回提供精製酵素サンプル製造)を行います。
Q:遺伝子組み換え技術を利用しない方法での酵素開発も可能?
A:可能です。詳細は、「digzyme Express」のご紹介ページ(https://www.digzyme.com/cms/wp-content/uploads/digzyme_Express_ol.pdf)をご参照ください。
Q:「digzyme Custom Enzyme Lab」は食品産業向けのみのソリューション?
A:「digzyme Custom Enzyme Lab」は、食品産業だけでなく、化学産業など業種を問わずご利用いただけるソリューションです。
Q:提供された精製酵素サンプルの中から、希望に合う酵素が見つかった場合、その後はどうなる?
A:「digzyme Custom Enzyme Lab」で開発された酵素については、シームレスに製造に向けた開発へ進めていくことが可能です。digzymeでは、該当酵素の製造技術開発・各種認可まで、お客様の開発テーマの事業化に最後まで伴走いたします。
Q:開発した酵素ライブラリの知財の扱いはどのようになっている?
A:digzyme Custom Enzyme Labで開発した酵素の中から有望な酵素を見出すことができ、更にそれを利用した事業化をご検討される際には、ご要望に応じて柔軟に対応させていただきます。
以上、「digzyme Custom Enzyme Lab」でご提供致しております内容についてご回答申し上げましたが、実際の開発の際には、お客様のご要望に応じて柔軟に対応させていただきますので、お気軽にご相談ください。
今回のQ&Aは以上となります。
最後までお読みいただき、ありがとうございました。
ご不明な点やご質問がありましたら、ぜひ下記のコンタクトフォームよりお気軽にお問い合わせください。
【▼コンタクトフォーム】
https://www.digzyme.com/contact/
糖鎖データベースからの目的単糖を含む多糖の検索と可視化
はじめに
digzymeにてインターン勤務させていただいております、東京大学修士2年の高栁龍です。大学では、タンパク質のリン酸化やタンパク質立体構造に関連した研究などを行っています。今回のtechblogでは、研究開発業務の一環として新たに開発しました、目的単糖を含む多糖の網羅的検索と可視化ツールであるGlycoSearcherを紹介いたします。
近年、デンプンや食物繊維で代表されるように、糖鎖など多糖の研究と産業利用が活発となってきています。新規の糖質を開発する需要が上がってきており、その中で多糖は構造の多様性が非常に高いものとして注目されています。そこで、様々な多糖を網羅的に検索するためのツールとして、新たにGlycoSearcherを開発いたしました。
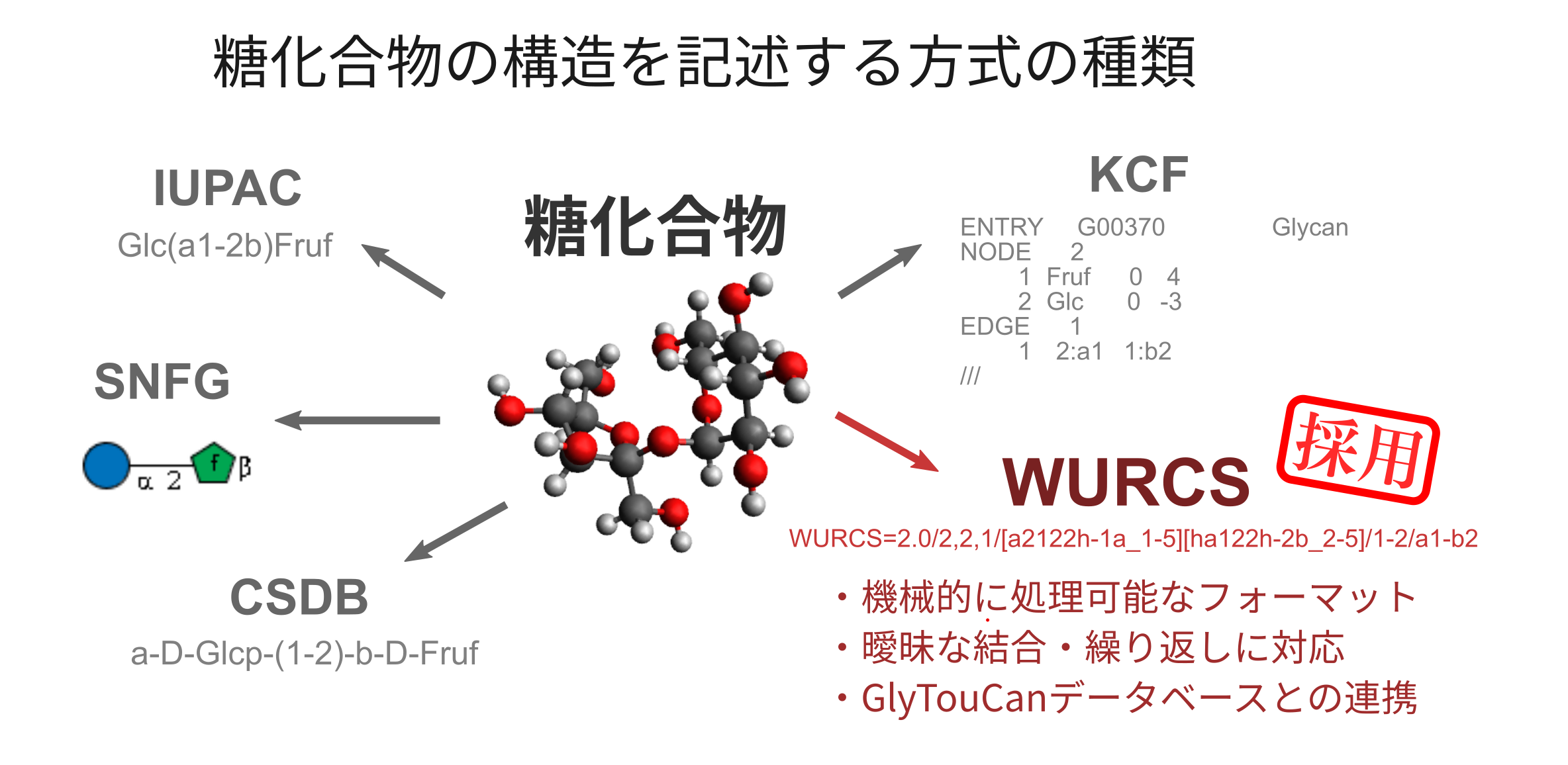
多糖の記述方式とデータベース
今回対象としている糖化合物は、既に数十万を超える数が報告されており、データベース化されています。こうした多くの糖化合物の中から目的に沿った多糖を厳選し、合成経路の探索や酵素開発などの応用につなげるには、計算処理しやすい記述方式と網羅性の高いデータベースが必要となります。
糖化合物の構造を記述する方式には様々なものが知られています(図1)。SNFGやKCFといった形式は可視化にすぐれていますが、構造情報の抽出や比較など応用的な計算処理には不向きです。一方、IUPACは、人と機械の両方が読み取り可能で簡潔な構造表現ですが、繰り返しなど複雑で曖昧な表現に対応するのは難しいです[1]。そこで、GlycoSearcherでは、計算処理に適し、繰り返し表現可能なWURCS形式、およびWURCS形式での糖化合物情報を収集したGlyTouCanデータベース[2]を採用しました。
(図1)
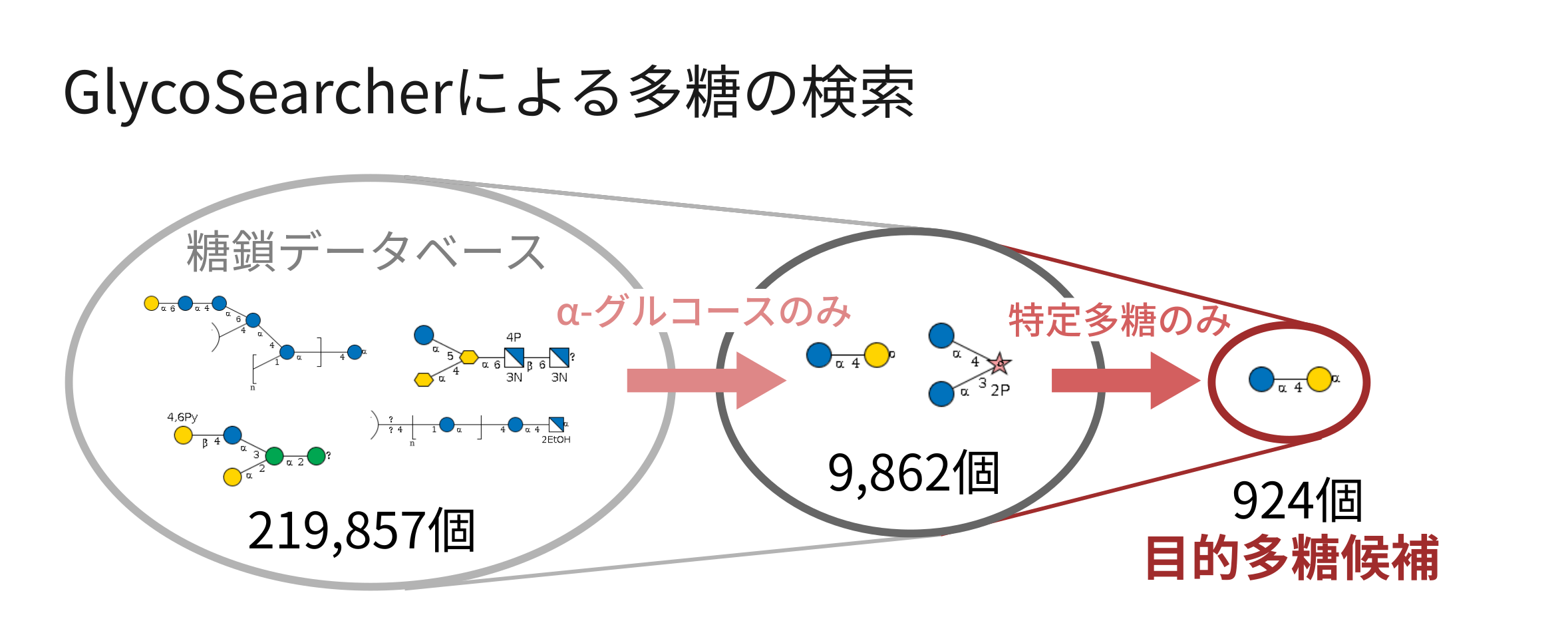
GlycoSearcherを用いた検索
ここにGlycoSearcherでは、膨大な量の候補から目的に沿った多糖を抽出することが可能です。例えば、グルコースやガラクトースなど、特定の単糖単位を含む多糖を検索することができます。さらに、多糖を構成する単糖単位を一部のものに限定する、フィルタリング機能も兼ね備えています。これにより、ある特定の単糖を原料とし、その他特定の糖のみを用いて合成可能な多糖を列挙することができます。
以下に、α-グルコースを例に実行した結果を紹介します(図2)。219,857個ある糖鎖の中から、α-グルコースを含む多糖を検索したところ、9,862個が検出されました。さらに、構成単糖をグルコース・ガラクトース・フルクトースのみに絞ったところ、924個まで候補が減少しました。
(図2)
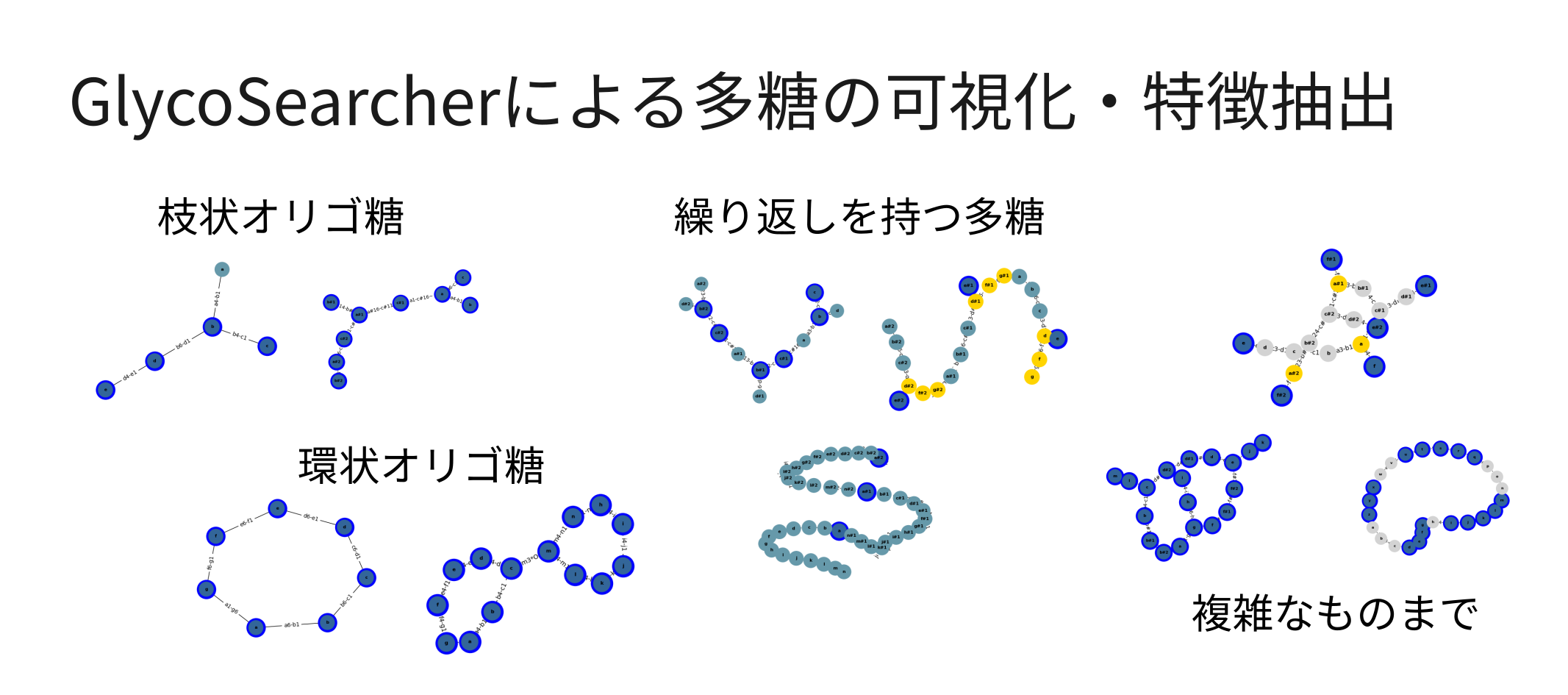
多糖構造の可視化と特徴抽出
得られた検索結果は、効果的に可視化され、続く応用処理へと活かすことができます(図3)。WURCS形式の多糖をグラフとして再構成することで、数千個の検索結果を数分のうちに描きだすなど、高速な可視化が可能です。また、曖昧な繰り返し回数を持つ構造についても、特定の回数だけ繰り返しを展開することで、実質的な構造を可視化するのみならず、曖昧なままでは難しい構造間の比較など、更なる計算処理へとスムーズに移行させることができます。
検索結果の多糖がグラフ化されていることから、多糖構造に対する特徴抽出も可能です。例えば、得られた多糖構造の末端にグルコース単位が存在しているか、あるいは特定の構造(モチーフ)を含むか、といった計算を行うことができます。さらに、検索によってヒットした多糖について、PubChem[3]などの各種データベースと統合させることで、その一般名や関連する酵素情報と統合させることができ、多糖を含む反応についての情報を得ることが可能です。
(図3)
終わりに
今回開発したGlycoSearcherでは、目的の多糖をデータベースから網羅的に検索し、更なる計算処理へと応用させることができました。さらに、得られた目的多糖候補から情報を抽出しその合成に関与すると予測される酵素情報を取得することで、その後の酵素デザインのワークフローにつなげる体制を整えられました。
謝辞
糖化合物に関する知識の習得を始めとしたGlycoSearcherの開発には、事業開発部の礒崎さんに大変お世話になりました。この場を借りて感謝申し上げます。
参考文献
[1] 細田 正恵, 木下 聖子.「糖鎖関連インフォマティクスへの入り口」 JSBi Bioinformatics Review, 2(1), 87-95 (2021).
[2] https://glytoucan.org/
[3] https://pubchem.ncbi.nlm.nih.gov/
構造予測とMDシミュレーションを用いた酵素活性予測の実用例
はじめに
事業開発部の礒崎です。弊社では有用酵素探索の1つとして、分子動力学シミュレーションを用いた酵素活性予測を行っています。構造未知の酵素配列からその構造を予測し、標的化合物との複合体を分子動力学シミュレーションへ供します。その結果から、digzyme独自のスコアを算出し酵素活性を予測します。本ブログでは、その具体例としてthiolaseの1つoleAという酵素の類似配列から実際に活性をもつものを予測した結果をご紹介します。
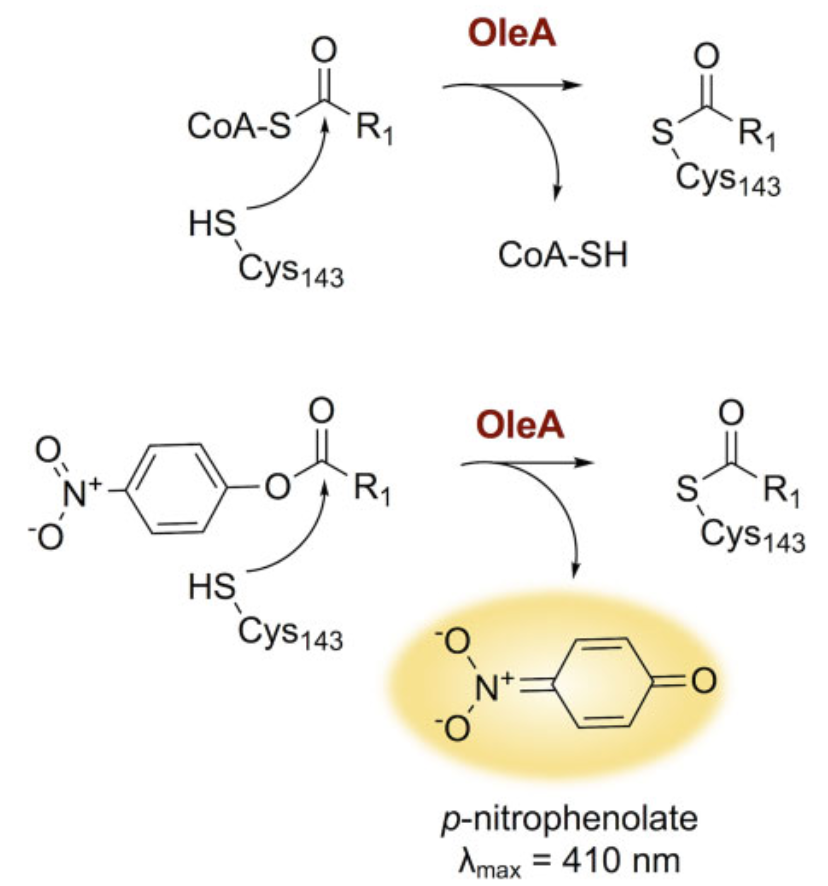
酵素活性予測に使った材料
oleAの本来の基質はacyl-CoAです。このアシル基をoleAのCys143が脱離させます。この活性を調べる上で、p-nitrophenolateを用いた実験系が使われます(図1)。
結果
oleA類似配列59配列を対象にp-nitrophenolateの1種4-nitrophenyl-hexanoateを加水分解するか予測しました。
1.類似配列59配列の3次元構造予測
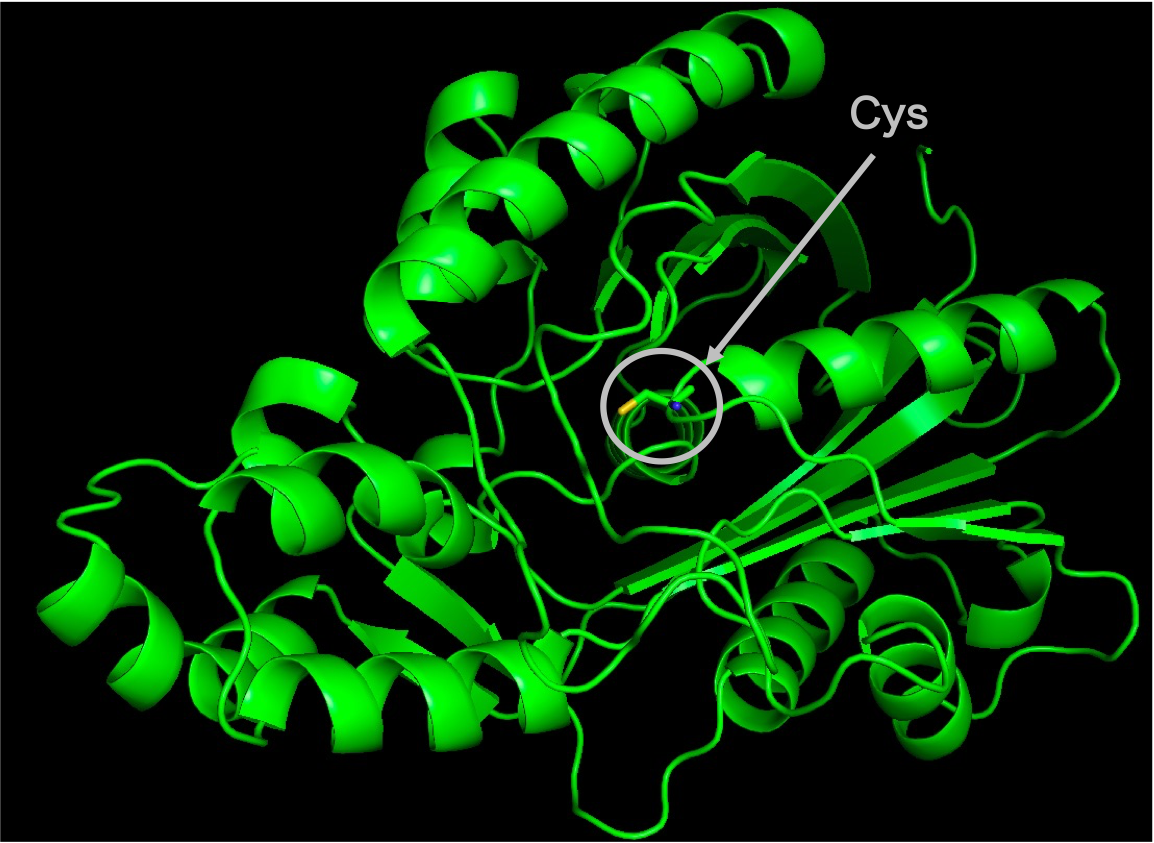
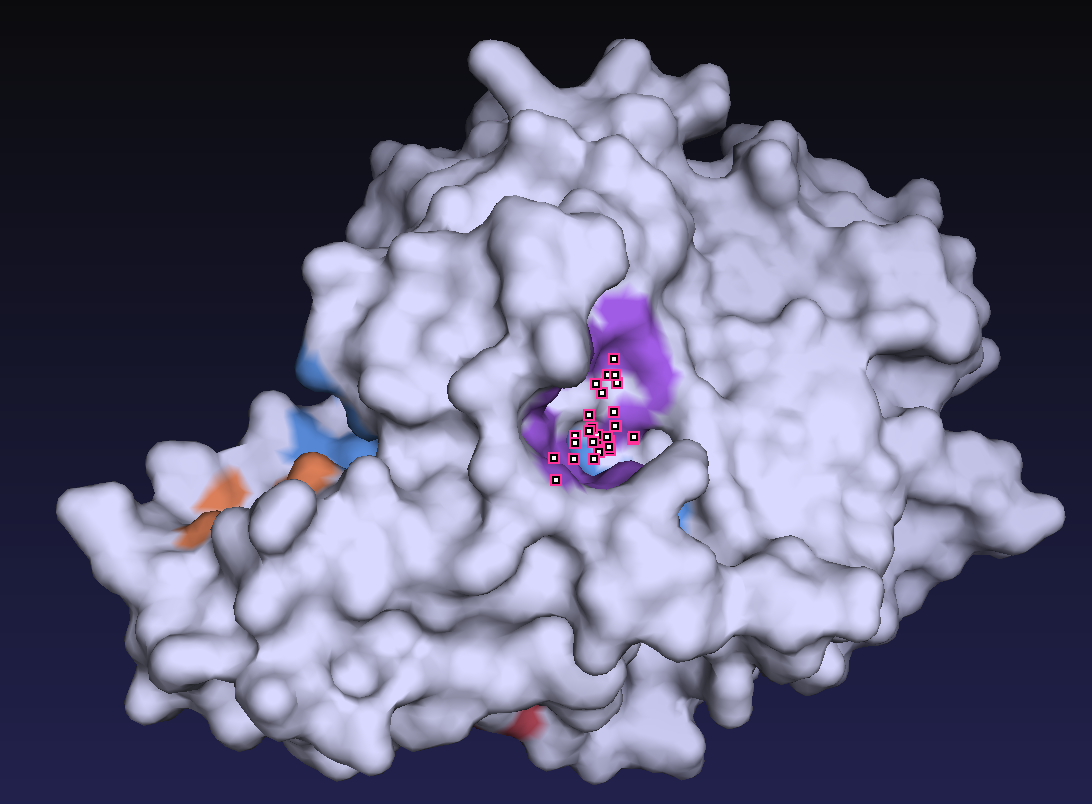
まずは類似配列59配列すべての構造が未知であるため、その3次元構造を予測し、予測した構造から活性残基の位置と基質が入るポケットの位置を予測しました。図2は類似配列の配列情報から予測した3次元構造および活性残基Cysの位置を示しています。図3が基質が結合するポケットの位置を予測した結果です。
2. 分子動力学シミュレーション
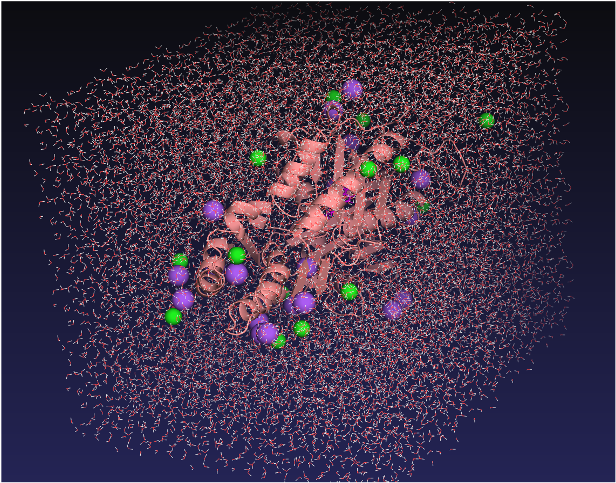
続いて、水分子とイオンの中に酵素と基質である4-nitrophenyl-hexanoateの複合体を配置して分子動力学シミュレーションを実行します(図4)。
3. digzyme独自酵素活性予測スコア算出
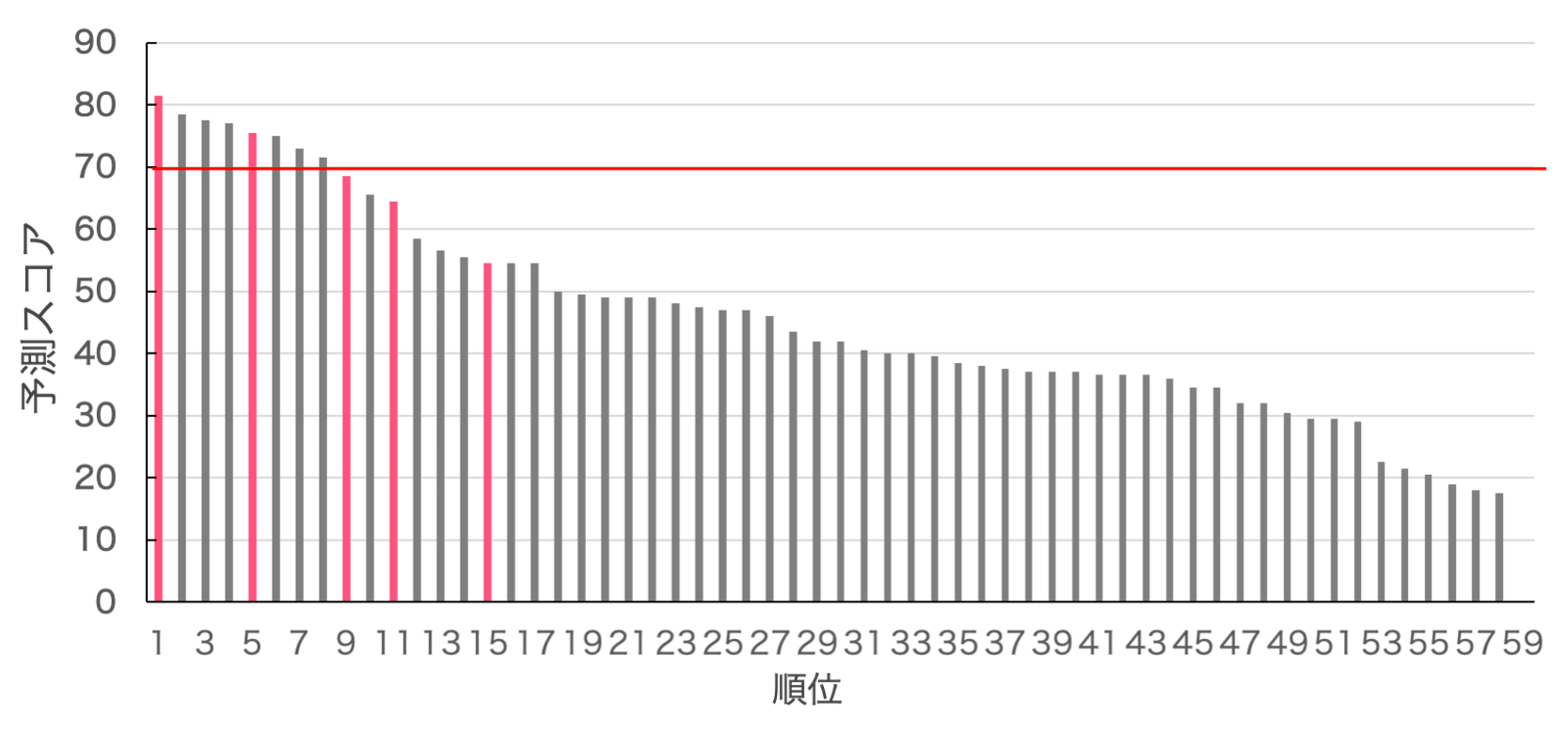
最後に分子動力学シミュレーションの結果から算出したdigzyme独自のスコアを計算します。図5にスコアが高い順に全59配列の予測スコアを記載しました。実証実験で活性があった配列をピンク色、活性がなかったものを灰色で表示しています。スコア70以上の配列を活性ありと判断しています(図5の赤線より上)。今回は9配列を活性ありと予測し、そのうち3配列が実際に活性を持っており、陽性適中率(PPV) = 0.30でした。また、真陽性率 (TPR) =0.6、偽陰性率 (FPR) =0.13という結果でした。このことから、不活性なものをランキング下位に分類できており、上位の配列に実際に活性のある配列を含んでいることが確認できました。
終わりに
本ブログでは、酵素活性予測技術を用いて、実験で活性が確認された酵素の活性予測を実演しました。通常実証実験を行う場合5~10配列を合成します。今回は上位5配列の中に実験で活性があった2配列が含まれており、弊社の酵素活性予測の精度が実用に適うものであると示されました。特に母集団の酵素配列のうち活性のあるものがわずかしか含まれていないようなケースを想定してデータを選びました(今回は59配列中5配列)。偽陰性率が低く抑えられているため、正しく酵素活性が予測できています。
謝辞
今回の酵素活性予測の材料として以下の論文から実証実験データを利用させていただきました。
Robinson et al., (2020) Machine learning-based prediction of activity and substrate specificity for OleA enzymes in the thiolase superfamily. Synthetic Biology.
