活用されずに眠る配列データの宝の山を探索:鈴木彦有が語る、価値を引き出す解析の現場(社員インタビュー)
はじめに
本記事は、弊社のnoteに2024年11月に掲載されたインタビュー記事を、より多くの方にご覧いただけるよう、当テックブログに転載したものです。内容は掲載当時のものとなります。
(※記事中の組織名・役職等はすべて取材時のものです。※文中では、社内での呼称に合わせ「彦有さん」と表記しています。)
本文
「インフォマティクス×酵素開発」
東京工業大学で分子進化を学び、ビッグデータ解析の専門家としてdigzymeに参加した鈴木彦有さん。
今回は、研究開発の裏側から見た「データ駆動の酵素探索」の現場についてお話を伺いました。

ーー早速なのですが、digzymeご入社の経緯を教えてください。
はい。具体的に転職を考え始める以前 からJREC-IN(科学技術振興機構が提供するキャリア支援ポータルサイト)に登録していたのですが、そこにdigzymeの求人が出ていたのをたまたま目にして。
母校の東工大発だし、縁がありそうだなと思ったのと、自分自身のスキルの評価として、僕が持っているノウハウが使えて即戦力になりそうだなと感じたことが応募のキッカケです。
ーーなるほど。前職ではどんなことをなされていたのですか?
ゲノムとかトランスクリプトーム・・・いわゆるオミックス解析と呼ばれる、バイオロジーのビッグデータを対象とした、解析サービスを提供している会社にいました。digzymeと似てるところは、オーダーメイドに近い案件が多いところでしたね。
ーーdigzymeでは共同研究として受けているような案件に近いでしょうか?
近いです。もちろん酵素を対象にした開発っていうわけではなかったんですけれど。
ビッグデータを使った解析って、計算機のノウハウが必要なので誰にでもできるわけでは無いので、食品関係や製薬関係の企業さんなどから頂いた案件に対してそれぞれに応じた解析を行い、なんらかの結論を返す、というお仕事をしていました。コンサル業のような形をイメージしていただくとわかりやすいかもしれません。
ーー研究開発のコンサルティングに、バイオのビッグデータを絡めていた?
そうですね。そういうベンチャー企業に5年くらいいました。扱う案件としてわかりやすい例だと、やっぱり抗体医薬品関係。近年の製薬会社の売上高上位を占める製品は抗体医薬品ですから、関連した解析をすることが多かったです。
「抗体」は、特定の抗原(ターゲット)だけに結合する能力を持ったタンパク質です。
なので、抗体医薬品は従来の医薬品より副作用も減らせますし、がんや難病の治療時に強力な効果を発揮するものなのですが・・・抗体は「タンパク質」の一種なので 、もちろんベースになる「ヒトの抗体の遺伝子」があるわけです。
この抗体遺伝子をハムスターの培養細胞に組み込んで、大量に作らせて精製して、製品にする、という流れがあるんですが、その過程で、培養方法をどう工夫すればより効率的に大量に作れるか・・・
あるいは、新しい抗体医薬品を作るとしたら、どういう配列のものを作ればいいか、などを考える必要があります。これらを考察する時に、ビッグデータから知見を得るということが最近ではよくあるんですね。
例えば、少量のスケールならできていたことを、実生産の大スケールで行おうとしても、少量の時の単なる比例にはならない。100倍のスケールにしたら100倍できるかっていうとそうはいかないので(笑)そういう時に何が起きているのかを分析したりするために、生き物のビッグデータを取得して調べるアプローチがあり、それらのお手伝いしていました。
ーーなるほど。量産時の生物のコントロールは難しいと、中村さんもおっしゃっていました。
digzymeに入社後はどのような業務に関わってきましたか?
入社直後は、合成生物学の発想に基づく開発がメインでして、目的化合物を生き物に合成させるための、新規の反応を含む反応経路の探索プログラムの開発をしていました。
例えば出発化合物Aから目的化合物Dを作りたい、というときにAからBを経て、Cを経て、Dを作らなければならないとします。途中に何段階かの反応を噛ませないといけないということは、この間を繋ぐ反応経路の推定をしなければ、それらの反応を触媒する酵素探索ができないわけです。
ちなみに合成生物学では培養細胞や微生物を使って、何か人間にとって有用な物質を作りたいとなった時に往々にして一反応では済まないので、反応経路の推定は不可欠なんですよね。
ということで、色々な経路がありえるなかで、最適なものを探索できるようにしていました。
最近のdigzymeは産業用酵素をメインに扱っているので、お客様の求める酵素そのものを作れば良いわけですから、反応経路を気にする必要はなくなっています。
こういう反応を触媒する酵素が欲しいです、という酵素を提供できれば良いので、酵素のバリエーションをライブラリとして持っておくための開発に注力していますね。
ーーライブラリの開発にも触れていただきありがとうございます!最近のお仕事も教えてください。
DRY解析に詳しくないWETのメンバーにも扱える解析環境の開発・保守をしています。
ーー具体的にはどのような内容になるのでしょうか?
一般的にIDE(Integrated Development Environment/統合開発環境)の導入とい言われるところですね。いざ解析を始めようとすると、初心者にとっては解析ソフトウェアのインストールだけでも高いハードルになります。皆さんなんとなく『インストーラーをダウンロードしてきてダブルクリックすると、コンピュータに入る』みたいなイメージをお持ちかと思うんですが、バイオインフォマティクスでよく使われる多くのツールって、そうじゃないんですよ。インストールの段階から全部コマンドラインで文字列を打って、コンピューターに命令を出していくので、初心者がいきなりやろうとすると、まずこのインストールでつまづいてしまうくらいコンピューターに関する専門知識がある程度必要だったりするんですね。なので、そのような工程が不要になるように、なるべく環境ごと用意して渡せるようにしていく・・・というのが別に弊社でなくても、よくある解決手段なのですが、IDEの導入というのはまさにそれのことです。
さらに、バイオインフォマティクスの多くのツールはGUI(Graphical User Interface)が提供されていないのでより親しみにくいのですが、直感的に操作できるところまで持っていくのは大変にしても、jupyter notebookを利用することでこれもできるだけ解消するようにしています。
これはどういうことかというと、WETの方も後からのトレース用に『実験ノート』を作りますが、同じように『解析のノートブック』を作るんです。もちろんただのメモじゃなくて、実行コマンドが書いてあるし、押すとそのコマンドが実行されるボタンもついている。このような機能を整備することで、同じようなコマンドラインを別の案件で流したい時に役立ちます。
極端な話、一個一個の処理にどういうプログラムが使われているのか判っていなくても解析ができちゃうんですよね。もちろん全くわかっていないで触るのは問題なので、あくまで極論を言えばですが(笑)
田村さん(※注1:インフォマティクススペシャリスト、田村康一さん)や礒崎さん(注2:プリンシパルインベスティゲータ、礒崎達大さん)とも適宜協力してこのような環境を作っております。
ーーなるほど。ちなみにWETの方々はどのようなシチュエーションでDRY解析をされるのでしょうか?
まず想定されるのは、お客様とNDAを締結した後で、蓋然性を検討するフェーズですね。
理想的には、WETの研究員とお客様が面談している最中にささっと解析をして蓋然性の検討ができると、DRYの研究員メンバーがその場にいなくても簡単なご返答ができてとてもスムーズですから。
また、実際の事業案件として研究開発を進めていくフェーズでも、DRY解析の結果に基づいて実験対象や方針決めをWETの研究員だけで完結できることは開発速度の面でメリットが大きいと思います。
ーー教えていただきありがとうございます。その他にはどのようなお仕事をなされていますか?
個別の案件で実際の解析業務や調査業務を担当したり、新たな解析アルゴリズムを考案することもあります。最新の論文を読んで、社内の既存のツールの精度を高めるための実装に使ったり、バイオロジカルな方面で、判定のための指標値の計算について考えたりということがありますね。
ーーどんな論文を読むことが多いですか?
もともと分子生物学、分子進化学が専門なので、タンパク質のアミノ酸配列などを調べていったり、ゲノムとか、トランススクリプトームとか言われる配列と紐づく「量」の情報を扱うような解析が得意なので、遺伝子配列を直接扱うような論文を読むことが多いです。
先ほど説明した『ノートブック』のなかで、配列データの処理としてうまく使えるところがないかなどを検討します。
ーー同じインフォマティクス・スペシャリストの田村さんは構造的な側面の専門家ですが、彦有さんは遺伝子配列の側面から日々解析を進めてくださっているんですよね。
そうですね。ちょうど得意分野が分かれているので、digzymeでは酵素を多角的な視点から捉えた解析が可能となっていて、そこが強みの一つかなと思います。
ーーでは次に、digzymeで働いていて、どんなところにやりがいを感じられるか教えてください!
やはり、開発した成果が直接、お客様にとっての新たな価値や当社にとっての利益を生み出すための手段として使われる可能性が高いところですね。
『世の中のニーズに応えられるかのテスト』という側面からのPoC後も、ずっと長くお付き合いくださっているお客様も多いです。秘密保持上多くは語れませんが、digzymeとのシナジー効果が大きいと感じていただけているのかな、と。
今後もニーズに対して新しい価値を提供して、貢献していきたい気持ちがあります。
ーー彦有さんは、digzymeだからこそできる新しい価値の提供についてどのようにお考えですか?
冒頭でお話した内容とも重複しますが、まず前提として、酵素に関わる様々な遺伝子の情報がビッグデータとして既に存在しています。その中には、配列は取られているけど機能がよくわかっていないものが大量にある。
配列を取っている=天然に存在している=判っていないだけで、何かしらの機能を持っている、ということで、人類にとって有用な反応を触媒できるような酵素がまだまだ沢山存在する可能性があるということ。それをどうやって探し当てるか?というときに、一個一個実験をして探していくのは到底無理ですので。最初に『この辺にありそうです』という絞り込む作業を計算機上で行い・・・もちろん最後は実験をしなければいけませんが、なるべく少ない実験のなかで当てられる確率が高くなるようにしていく。ですから最初に絞り込む作業ってすごく重要ですし、digzymeの技術の本質はそこにあります。
我々が掲げる『世界を変える酵素を、迎えにいこう。』のミッション通り、(注3:2024年11月時点。現在は「ものづくりに、まだ見ぬ選択肢を。人と地球に、持続する活力を。」に変わりました。)これからもビッグデータのなかにある宝の山を発掘して、迎えに行きたいですね。
ーーdigzymeの技術の本質、掲げるミッションに関しても触れていただきありがとうございます!
ちなみになのですが、配列は取られているけど機能がよくわからないものが、ビッグデータ上に大量に登録されている背景に関しても教えていただけますか?
もともとは個々の研究者たちが研究している遺伝子の配列を登録することに始まり、それをあとで第三者が検証したり、検証とは別に、他の研究に再利用して使えるようにしていたんですね。ところが、DNAの塩基配列を解読するシーケンスの技術が飛躍的に向上したため事情が一気に変わってしまいました。特に2010年代からは次世代シーケンサーと呼ばれる解読装置が普及 してきてしまったため、それまでとはうって変わって、安価に大量の塩基配列を解読できるようになりました。なので、今までは目的の遺伝子だけを狙ってPCR法などで増やしてから塩基配列を読むのが普通だったんですけれど、そうやって特定の遺伝子を増やして読むのではなくて、『ゲノム全体を読んじゃいましょう!』とか『転写されているRNAを全て読みましょう!』といった感じで『とりあえず全部読む!』ということができちゃう時代に突入してしまったわけです。
そうすると大量に読みまくったデータが、どんどん溜まり続けていくんですよね、いろんな生き物で。
これらに関して、もちろん別の研究に流用できる期待値が高いデータだという側面はあるものの、データを取った人たちは結局、自分の研究に関わる部分しか見ない、というか見れないことが多いです。
一方で、長年研究をしていると、別の研究で取られたあるデータについて、自分の研究にとって有用かどうかという視点で見てると『使える』ということはよくあります。
なので、digzymeも研究者ならではの視点でデータの再利用性に着目するという発想に辿り着きました。これからも、まだ開拓されていないニーズに対して、将来的に応えられる可能性があるデータを埋もれているところから掘り起こす企業活動をしていきます。
ーーなるほど。背景についてもよく理解できました。様々な知見をお持ちの彦有さんですが、大学ではどのようなことを専門になされていたのでしょうか?ぜひ改めてお聞かせください。
学生時代は、東京工業大学(現・東京科学大学)の岡田典弘先生(東京工業大学名誉教授)と二階堂雅人先生(東京科学大学准教授)のもとで、進化についての研究を行っていました。
具体的には生物の遺伝子やタンパク質といった分子レベルのデータを使って、異なる生物間で遺伝子の進化的な系統関係を調べたり、そこから発展して遺伝子がどのように進化するか、また祖先の生物が持っていた遺伝子の機能を考察したりしていました。
僕が配属された当時、岡田研究室では東アフリカの三大湖(ビクトリア湖、マラウィ湖、タンガニイカ湖)に生息するカワスズメ科の淡水魚(シクリッド)に着目していたのですが、この魚は形態的・生態的に多様性に富んでおり、それぞれの湖に固有の種が数多く生息していることから生物進化や種分化(生物の種が分かれていくこと)を考える上でのモデルとして世界的にも研究対象になっていました。シクリッドが種分化を繰り返しながら莫大な多様性を獲得するに至った過程について、遺伝子から説明しようというチャレンジな研究が、研究室の内外で精力的に進められていたんですね。
この頃、同じく岡田研にいらっしゃった寺井洋平先生(総合研究大学院大学准教授)らのチームが行ったシクリッドの視覚関連遺伝子に関する研究などから、感覚に関係した遺伝子が先に進化することで種分化が促進されるという感覚駆動(sensory drive)仮説が提唱されており、これを受けて視覚以外の他の感覚についても感覚駆動仮説を検証しようと、二階堂先生のチームでは嗅覚に着目した研究を進めていました。
僕はそのなかでも特に、外界にある様々な匂いの物質をキャッチしてその刺激を細胞に伝えている嗅覚受容体遺伝子に着目し、その進化について調べていましたね。
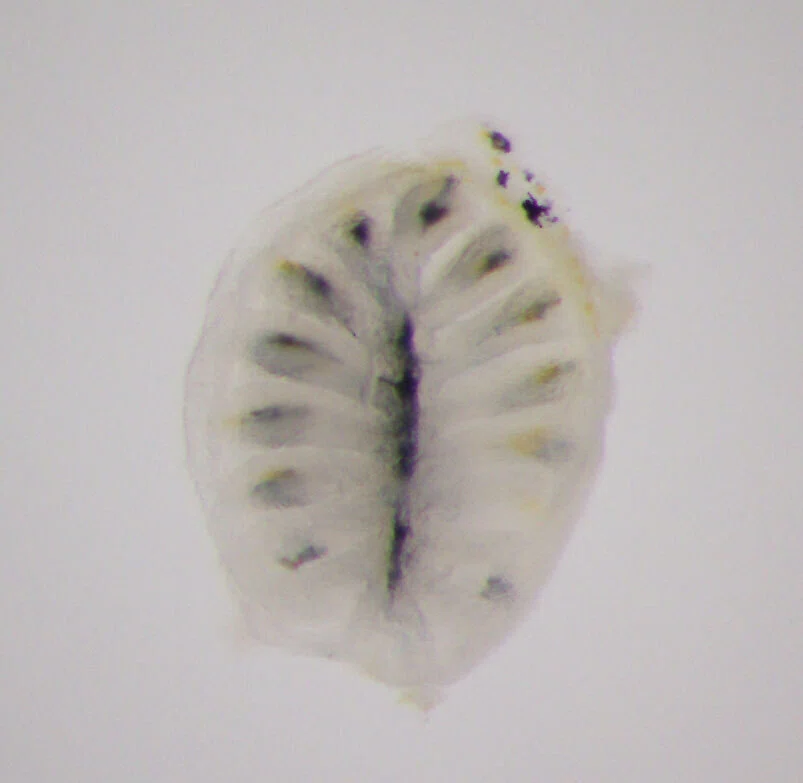
魚の鼻の穴を切り開くと、嗅房(olfactory rosette)と呼ばれるひだが沢山ある花びらのような構造体を肉眼でも見ることができるのですが、実はそこに匂いを感じる嗅神経細胞が多数集まっていて、魚が水の中の匂いを感じるための器官となっています。
これらの嗅神経細胞は一つ一つが別々の嗅覚受容体遺伝子を発現していると考えられているのですが、多数の嗅覚受容体遺伝子を分子系統学的に分類し、系統ごとに発現している嗅神経細胞を可視化したり、嗅覚受容体遺伝子以外にも嗅神経細胞に発現する嗅覚マーカータンパク質を使って、それらが発現している嗅神経細胞を可視化するといったWET実験に取り組んでいました。
そういったことに取り組むうちに、シクリッドに関する研究テーマからは少し離れて嗅覚受容体遺伝子や嗅覚関連遺伝子そのものの進化を調べていく方向に研究がシフトしていった感じです。
いくつか研究成果を紹介させていただきますと、ゲノムデータの解析から多くの脊椎動物は1つしか持たない嗅覚マーカータンパク質の遺伝子を真骨魚類は2つ持つことを二階堂先生が発見していたのですが、僕がその2つの遺伝子の発現を細かく調べたところ、それぞれ発現している嗅神経細胞が異なっており、さらに1つは嗅神経細胞以外に目の中の網膜の水平細胞にも発現していることが明らかになりました。
それまで嗅神経細胞のみに固有の発現を示すと思われていた遺伝子が、実は網膜の神経細胞でも使われていることがわかったのは面白いと思います。
また、シーラカンスのゲノムデータの解析に取り組んだ際、従来は陸上動物型と考えられていた嗅覚受容体遺伝子のいくつかの系統群について、実はシーラカンスやポリプテルスといったいわゆる「古代魚」と呼ばれる古いタイプの魚類にも数十個程度は存在することや、古代魚を含めた脊椎動物全般が保持している未知のⅠ型フェロモン受容体遺伝子(ancV1R)を発見したりもしました。
ancV1Rの発見については論文が出版された際、東工大のプレスリリースでも公表させて頂きまして、自分の中で特に思い入れのある研究です。

そんななか、あるときから次世代シーケンサー(数千億から1兆にまでのぼるDNAの塩基配列を短時間で解読する装置)から出力されるデータを用いた解析も行うようになりました。それにはいくつかきっかけがあったのですが。
もともと僕がいた岡田研はゲノムや個々の遺伝子の 配列を調べる解析を用いることが多く、バイオインフォマティクスを独学で学んで研究に使っている助教の先生やポスドクの先輩も何人かいらっしゃったので、じゃあ僕もちょっとやってみよう、と。
ーー独学で。
はい。コンピュータのプログラミングは、小さい頃に『BASIC』を遊びで触っていた経験もあったので、文法が似ているプログラミング言語のPerlを使い始めました。今ではあまり使われなくなってしまった言語ですが、当時はWebアプリケーション開発でもよく使われていて、文字列処理を得意とする言語ということもあり、塩基やアミノ酸の配列データ主に扱うバイオインフォマティクスにはうってつけでした。
そういうことをしているうちに、研究室では「バイオインフォマティクスができる学生」として認識されていきました。
その後、長谷部光泰先生(基礎生物学研究所教授)を代表とする「複合適応形質進化の遺伝子基盤解明」(2010年度から2015年度まで)という文部科学省の新学術領域研究公募テーマに岡田研が提案した課題が採択されまして、その際「バイオインフォマティクスができる学生」として岡田先生や二階堂先生の推薦もあって、ありがたいことに僕も参加させて頂けることになりました。
このプロジェクトは、当時普及し始めていた次世代シーケンサーを積極的に用いてゲノムやトランスクリプトームといったバイオのビッグデータを実際に取得して活用することで、あらゆる生物の複雑な進化現象の一端を解き明かそうというチャレンジングなテーマに挑戦するものでした。
それとともに、僕のような若手研究者に次世代シーケンサーのデータを扱うノウハウも含めたバイオインフォマティクスの技術を教授するといった教育的な側面もあったと思います。
実際、このプロジェクトに参加させて頂いたおかげで、これらの経験やスキルを積むことができましたし、それが前職そしてdigzymeの仕事でも活かされていると思います。
ーーなるほど。詳しく教えていただきありがとうございます。
WET,DRY両方に深く関わってこられた彦有さんですが、digzymeのなかでは今後どのようなことにチャレンジしていきたいですか?
まさに、WET &DRY二刀流の人材育成ですね。WET側がDRYを知ることで研究の速度が向上したり、DRY側がWETを知っていることで生命科学の現場に合った開発ができるようになると期待されます。
実は前職でも、バイオインフォマティクスの解析技術のハンズオンセミナーに講師として呼ばれて、企業やアカデミアの方に向けての技術解説をしていた経験があるので。
digzymeでも社内セミナーのようなものができたらいいなというイメージを持っています。
ーー社内セミナー、いいですね!
はい。IDEを作ったのも布石になっています。
もちろんIDEを使用するだけではあまりノウハウにはなりませんが、今後はWETの新人さんが入社したタイミングで、ファーストステップとしての『IDEを用いた解析をやってみよう!』というようなセミナーも開けたらな、と。このあたりはCTOの中村さんとも相談して進めていけたらなと考えています。
ーーDRYの技術者にバイオ出身者が多いdigzymeだからこそできる教育ですね。そういった意味でも人材の揃い方は希少価値が高いですよね。
そうなんです。
digzymeはむしろ、純粋な情報科学の人は採用していないんです。なぜならそれだと『酵素』の開発はできないから。
渡来さん、中村さん、田村さん、僕、みんなバイオに関係する分野をバックグラウンドとしており、バイオ側から情報科学の知識を仕入れていった経験があります。
WETの皆さんはもちろん、今バイオの領域にいらっしゃるわけですから、僕らのようにバイオ側から情報科学を取り込むという方向性に進むことが可能ですし、その育成ができるのがdigzymeだと思っています。
本来こういった人材育成は日本の国策としても必要なので、業界全体で解決すべき内容だと僕は考えています。ですが、現実的に、WET出身者がDRYの技術を学ぶのって結構大変なんですよね。
一番の理由は、必要に迫られる機会が少ないからだと感じており、それこそdigzymeのように『バイオと情報科学両方がわかっていないと、開発が進まない環境』にでもいない限りなかなか難しいはずです。
ーー環境に左右されやすい?
といった側面もあります。
むしろ実験中心の研究室だと、実験もせずにパソコンに向かってカタカタしているだけだと遊んでいると見なされるというような話も聞いたことがありますし。
でも実際は、情報科学の人があえてバイオを勉強しないのはわかるんですけど、バイオの人は本当は情報科学とは切り離せないんですよ。
次世代シーケンサーで大量に配列を読むことが当たり前の時代になったので、いつまでもそれを避けているというのは、バイオの研究においてディスアドバンテージでしかないんです。
DRY部分は共同研究先にお任せすればいいという考えもありますが、そうするとバイオインフォマティクスができる先生方がとても忙しくなってしまうので(笑)
アカデミアでも本当は、各研究室でその生物を研究している方が自らDRY解析もできた方がいいと思っているのですが、これについては先ほどもお話しした進学術領域研究でコアメンバーだった先生方も当時から同じような理想を掲げておられたと思いますし、CEOの渡来さんも同じ意見だと思います。
ーー最後に、digzymeに応募を考えている未来の仲間に一言あれば、お願いします。
自身の探究心と他者への貢献の意志を両方持ちながら、仕事のできる環境があります。
ーー彦有さん、ありがとうございました。
終わりに
▼オリジナル記事はこちら(note)
https://note.com/digzyme/n/n7fbee270334b



