ifia JAPAN 2025会場で頂戴したご質問への回答まとめ
はじめに
食品事業部の村瀬です。
弊社は、2025年5月21日(水)〜23日(金)に東京ビッグサイトで開催された「ifia JAPAN 2025 第30回 国際食品素材/添加物展・会議」(主催:食品化学新聞社)に、昨年に続いて出展いたしました。
会期中は、弊社の技術に高い関心をお持ちの多くの来場者の方々と、直接お話しできる貴重な機会となりました。
出展ブースでは、そうした皆さまに向けて、弊社の最新の取り組みをご紹介していましたが、なかでも大きな目玉となっていたのが、新しいソリューション「digzyme Custom Enzyme Lab」のローンチについてです。(詳しくはプレスリリースをご覧ください:https://prtimes.jp/main/html/rd/p/000000018.000050097.html)


ローンチにあたり、想定を上回る多くのご反響をいただき、出展ブースでは連日、多くの方から具体的なご質問やご相談をいただき、終始活発な対話の場となりました。
そこで今回のテックブログでは、「digzyme Custom Enzyme Lab」のローンチを記念した特別編として、会期中にいただいたご質問の中から、特に多かった内容をピックアップし、回答と合わせてQ&A形式でご紹介します。
本ソリューションにご興味をお持ちの方はもちろん、「酵素を使った開発に関心はあるけれど、何から始めたらよいのかわからない」という方にも、ヒントとなる内容です。ぜひ最後までご覧ください。
Q:「digzyme Custom Enzyme Lab」は、どんな開発テーマに利用できる?
A:「digzyme Custom Enzyme Lab」は、「現行の酵素を用いた製造法の効率向上」のような具体的な開発テーマから、「(酵素を用いた)新規食品素材開発」のような抽象度の高い開発テーマにもご利用いただけるソリューションとなっております。精製酵素サンプル提供とお客様での酵素評価のフィードバックを繰り返し行うことで、随時開発方針の調整が可能です。
Q:提供される精製酵素サンプルについて、どんな情報が提供される?
A:基本的な酵素活性の有無、プロファイル(至適温度、至適pH、熱安定性、pH安定性)の確認を弊社で行い、サンプル提供と併せて提出いたします。実際の評価系での実証実験については、お客様ご自身で確認することができます。
Q:提供される精製酵素のサンプル量はどれくらい?
A:開発テーマごとにもご相談となりますが、典型的には、酵素溶液として~mL、タンパク質として~mg単位でのご提供となります。
Q:初期開発期間はどのように決まる?
A:お客様がご希望するテーマについて、事前に弊社で開発方法の検討・調査を行い、初期開発期間を決定いたします。基本的には、2〜6ヶ月の間で初期開発(in silico酵素デザイン〜初回提供精製酵素サンプル製造)を行います。
Q:遺伝子組み換え技術を利用しない方法での酵素開発も可能?
A:可能です。詳細は、「digzyme Express」のご紹介ページ(https://www.digzyme.com/cms/wp-content/uploads/digzyme_Express_ol.pdf)をご参照ください。
Q:「digzyme Custom Enzyme Lab」は食品産業向けのみのソリューション?
A:「digzyme Custom Enzyme Lab」は、食品産業だけでなく、化学産業など業種を問わずご利用いただけるソリューションです。
Q:提供された精製酵素サンプルの中から、希望に合う酵素が見つかった場合、その後はどうなる?
A:「digzyme Custom Enzyme Lab」で開発された酵素については、シームレスに製造に向けた開発へ進めていくことが可能です。digzymeでは、該当酵素の製造技術開発・各種認可まで、お客様の開発テーマの事業化に最後まで伴走いたします。
Q:開発した酵素ライブラリの知財の扱いはどのようになっている?
A:digzyme Custom Enzyme Labで開発した酵素の中から有望な酵素を見出すことができ、更にそれを利用した事業化をご検討される際には、ご要望に応じて柔軟に対応させていただきます。
以上、「digzyme Custom Enzyme Lab」でご提供致しております内容についてご回答申し上げましたが、実際の開発の際には、お客様のご要望に応じて柔軟に対応させていただきますので、お気軽にご相談ください。
今回のQ&Aは以上となります。
最後までお読みいただき、ありがとうございました。
ご不明な点やご質問がありましたら、ぜひ下記のコンタクトフォームよりお気軽にお問い合わせください。
【▼コンタクトフォーム】
https://www.digzyme.com/contact/
計算による酵素の熱安定性の予測
田村 康一(研究開発部)
NOVEMBER 2024
はじめに
酵素は多様な化学反応を触媒する生体高分子(主にタンパク質)であり、金属触媒に比べて環境負荷が低く、高い選択性を持つなどの優れた特徴を持つことから、工業・食品などの様々な用途で用いられています。一般に、化学反応速度は温度に依存し、温度が上昇すると反応速度も増加します。これは加温によって反応の活性障壁を乗り越えるためのエネルギーが供与されるためです。例として、無機鉄系触媒を用いて窒素と水素からアンモニアを合成する反応であるハーバー・ボッシュ法では、温度が 500℃ 近くに設定されるようです。一方で、生体高分子である酵素はそのような苛烈な条件に耐えることができず、ある温度でアンフォールド(unfold)し壊れます。この温度を melting temperature (Tm) と呼びます。Tm は、酵素の熱安定性を評価するための重要な指標であり、これを予測しさらに改善することがタンパク質工学における重要な課題となっています。digzyme では、より幅広いニーズに応えるために、酵素の Tm に相関するスコアを計算で予測する手法を開発し、高温で機能する酵素の選抜や、酵素機能の改良に用いています。本ブログでは、実験から得られたタンパク質の熱安定性に関する 2 つのデータセットを用いて、開発した方法の性能を評価します。
方法論
一般に、酵素探索や酵素改変の文脈で熱安定性予測の対象となる酵素は、由来する生物種と所属するタンパク質ファミリーが多岐にわたるので、予測モデルにはこれら2つの要素に依存しない汎用性が求められます。この要請を満たすためにこれまで様々な方法が提案されてきており、それらは次の 2 つに大別されます。
1.データ駆動型アプローチ
2.物理ベースモデルによるアプローチ
1.は、主に機械学習と呼ばれるアプローチで、実験によって得られた大量のデータから、タンパク質の熱安定性を予測するための法則(モデル)を構築します。このモデル構築過程を訓練と呼びます。訓練に要する時間は、データの量やモデルの複雑さに依存し、コーヒー1杯を飲んでる間に終わることもあれば、最新の GPGPU (計算を加速するための装置)を用いて1週間丸々かける場合もあります。いったん訓練済みモデルを構築すれば、その後の予測のステップの計算コストは訓練過程に比べて低いです。
そのモデル構築の方法から推察されるように、機械学習モデルの精度は学習データの量と質に大きく依存します。学習データが少ないと、見出される法則性にバイアスがかかることは明らかです。また、学習データが特定の生物種やタンパク質ファミリーに偏っていないか注意する必要もあります。さらに、タンパク質の物性値を測定するときの実験条件も揃っていることが望ましいですが、これは様々な研究グループからの実験データがデータベースに蓄積される現状を考慮すると、実現は困難であると言えるでしょう。
学習データの量と偏りに注意したとしても、依然として汎用性に問題が残る場合があります。それが過学習と外挿性の問題です。過学習とは、機械学習モデルが学習データに過剰に適合している状態を指します。この状態のモデルは、訓練に使用したデータに対しては非常に優れた予測精度を与えますが、それ以外のデータに対しては全く無力です。過学習を防ぐには、モデルの複雑さを適切に制御するなどの専門的な技術が必要となります。外挿性とは、学習データがカバーする領域外のデータに対する有効性のことです。タンパク質の文脈でいえば、学習データに似ていない(相同性が低い)新規なアミノ酸配列に対しても予測の信頼性を担保できる能力を指します。これは、酵素探索や改変においてクリティカルな問題になります。というのも、大抵の場合、物性値を予測したい新規酵素の既知配列(論文に物性値が報告されているようなアミノ酸配列)に対する相同性は低いからです。このように重要な能力である外挿性ですが、単純な線形モデル以外では、それを内在的に組み込んだモデルを構築することは困難です。従って、ここでのベストプラクティスは、「構築した機械学習モデルの適用範囲をよく理解して使うこと」となります。
2.の物理ベースモデルでは、原子間相互作用を記述するエネルギー関数の構築が予測モデル構成の出発点になります。原子・分子のような極微の世界を支配する普遍的な自然法則の数学的表現(方程式)は 100 年程度前からすでに知られており、最も簡単な例でその方程式を手で解き、実験値と比較することで、その予測の精確さを実感することができます(気になる人は、大学初年級の物理化学の教科書を参照してください)。この方程式は(光速やプランク定数などの)物理定数以外の人為的なパラメータを一切含まないので、適用範囲に普遍性があります。しかし、我々が興味を持つような多原子系(=酵素+溶媒)ではこの方程式は非常に複雑になり、最先端の計算機を用いても現実的な時間内に計算が終わることはないでしょう。そのため、実際には様々な近似を導入し、精確さと普遍性を代償にすることで方程式を簡単化し計算が可能な範囲に落とし込みます。この近似手法が、構築した物理モデルの精確さと適用可能範囲を定義することになります。近似手法として頻繁に用いられるものとしては、
●分子力学ベースの経験的エネルギー関数の導入
●統計力学理論による溶媒の取り扱い
があります。前者では、簡単な関数形を仮定し、人為的なパラメータセットを導入することで、本来複雑な原子間相互作用を(かなり)簡単に記述します。後者では、酵素の周囲に存在する多数の溶媒分子を明示的に表現することはやめて、ある種の平均的な量で置き換えてしまいます。これらの大胆な近似は計算コストを大幅に削減してくれますが、先に述べたようにその精確さと普遍性は損なわれています。
digzyme score
digzymeでは、酵素の熱安定性を予測するために、物理ベースモデルを採用しています。このモデルでは、酵素の立体構造情報を入力すると、あるスコア(とりあえず ”digzyme score” と呼びます)を出力します。このスコアは酵素の Tm と相関があるように設計されており、通常 1.0 付近の値をとります。回帰モデルではないので、 Tm そのものの値を予測するわけではありませんが、普通は複数酵素間の安定性の差だけを知れれば十分なので、このような仕様になっています。
事例 1. 変異体の熱安定性の予測
酵素の有用変異体を計算で設計し選抜するためには、多数の候補変異体の各種プロパティを計算し、それらの値に基づいて変異体をランク付けする必要があります。プロパティの中でも重要視されるのが熱安定性です。以下では、変異体の熱安定性予測のコンテストにおける複数グループと digzyme の結果を比較します。
鉄-硫黄クラスターの再生に関わる酵素である frataxin の 8 つの変異体について、野生型と変異体のアンフォールディング自由エネルギーの差(ΔΔGu)を計算で予測するコンテストが 2018 年に実施され[1]、その結果が 2019 年に論文として出版されました[2]。アンフォールディング自由エネルギー(ΔGu)は、式 (1) で示される、
ΔGu = Gu - Gf (1)
酵素のアンフォールディングに付随する自由エネルギー変化と定義されます。ここで Gu, Gf はそれぞれアンフォールド状態とフォールド状態の自由エネルギーです。一般に、酵素はフォールド状態の方が安定なので、ΔGu > 0 となります。ΔGu が大きいほど、酵素はアンフォールドし難い(=安定である)ことに注意してください。野生型と変異体のそれぞれについて、式(1) で定義されるアンフォールディング自由エネルギーを測定し、その差をとることで、変異によるアンフォールディング自由エネルギーの変化が以下のように計算できます:
ΔΔGu = ΔGu(変異体) - ΔGu(野生型) (2)
ΔΔGu < 0 ならば、変異によって酵素が不安定化したことになります。
Figure 1左に、 digzyme が計算した予測スコア(”digzyme score”)と実験値を示します。ピアソン相関係数は 0.87 でした。 これは既存の物理ベースの手法として popular な FoldX よりも僅かに良い結果です(Figure 1右)。
他の物理ベースの手法としては、 Pal Lab のグループが分子動力学法(molecular dynamics, MD)ベースの方法による予測を行っています。この方法では、 MD による構造サンプリング(1 ns)を行い、サンプリングされた構造を folded state と unfolded state にクラスタリングしています。その後、それぞれのクラスターの代表構造に対してエネルギー計算を行い、式(1)の値が算出されます。この計算を野生型と変異体について実行することで、式(2)から実験値との比較が可能な値を得ることができます。しかし、MD 開始構造である folded state のタンパク質が、この計算条件(27℃、通常の平衡 MD)にて 1 ns 以内に unfolded state に構造遷移するという前提には明らかに無理があります。というのも、fast-folding protein と呼ばれる、比較的短時間でフォールド/アンフォールドするタンパク質の Tm 付近でのシミュレーションにおいてさえ、構造遷移が生じるのにこの 1000 倍以上の時間がかかるからです[3]。したがって、この方法でも中程度の相関が存在した(Figure 1右)のは、偶然の結果と言っても過言ではないでしょう。
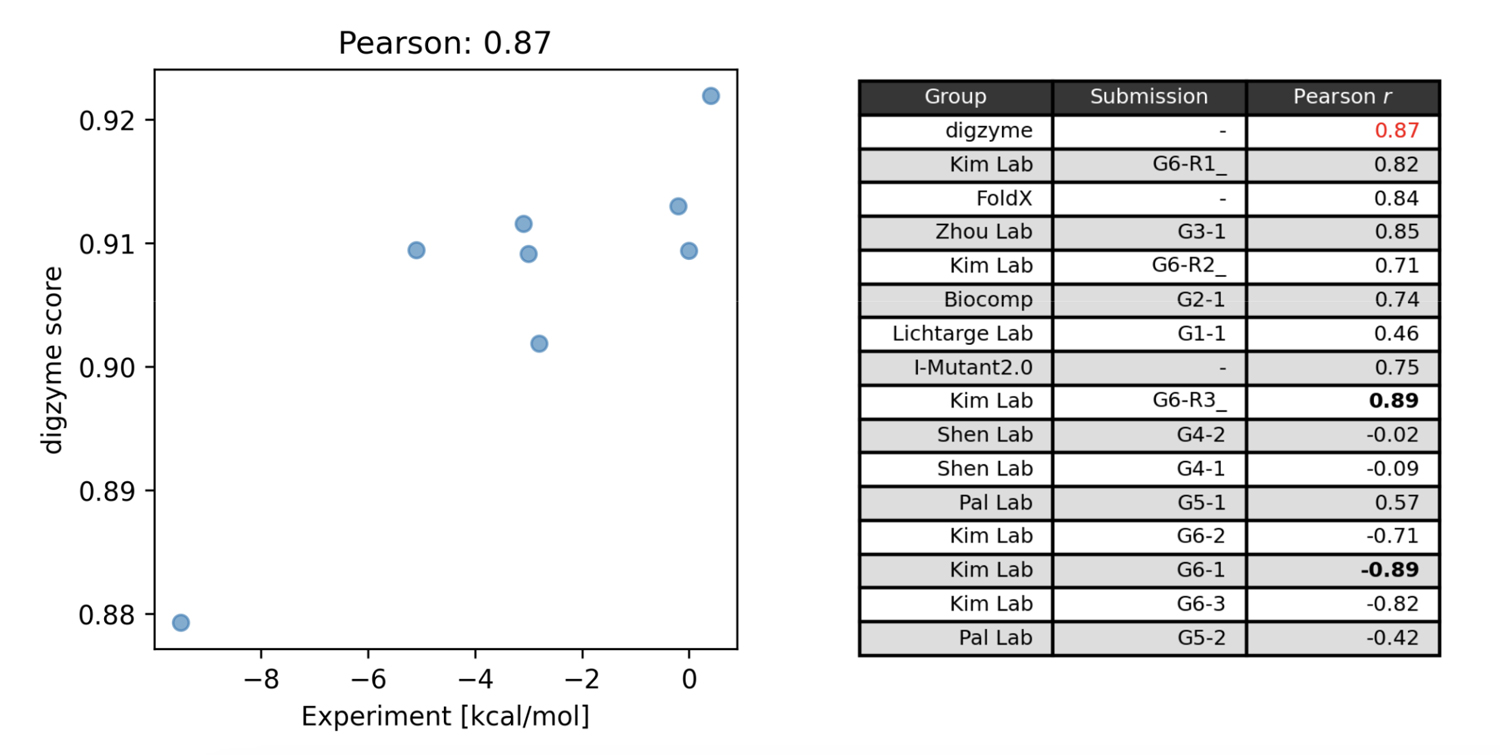
コンテストで最も相関の大きいモデルを提案したのは Kim Lab のグループで、相関係数の絶対値は 0.89 でした(Figure 1右)。このグループは機械学習モデルでの予測を行っており、Protherm データベースに登録されている変異体の熱安定性のデータを学習に利用しています[4]。この機械学習モデルでは、構造ベースとアミノ酸配列ベースの特徴量がそれぞれ計算され、これらの特徴量を用いて勾配ブースティング木によって回帰モデルが構築されます。構造ベースの特徴量の中には物理モデルである FoldX の計算値が含まれており、この値が最も重要な特徴量であることが知られています[4]。この事実は、FoldX の予測と同程度の精度を有する物理モデルの digzyme score を使用することで、さらに高度な機械学習モデルを構築できる可能性を示唆しています。目的の酵素について、大量の変異体データが存在する場合は、専用のモデルの構築を試みても良いかもしれません。
事例 2. 同一機能酵素の熱安定性の予測
有用酵素の酵素探索では、同一機能(と予測される)酵素のアミノ酸配列をデータベースから多数抽出し(母集団の形成)、それらを何らかの指標でランキングすることで候補酵素を選定します。変異体設計と同様に、このときも熱安定性が重要な指標の1つになります。
ここでの熱安定性の予測は、事例 1.でみたような野生型と変異体の差の予測とは趣が異なることに注意してください。通常、野生型と変異体ではアミノ酸配列の長さは同じであり、配列一致度は 99% に近いことが多いです。これに対し、特定の機能に絞ってデータベースから抽出した配列の母集団では、アミノ酸配列の長さはまちまちであり、互いの一致度は低いことが一般的です。このような集団に対して熱安定性を予測し配列のランキングを作成することは、以下に示すように困難である場合が多いです。
ここでの事例は、2024 年の 9 月にプレプリントサーバーに投稿された、ナノボディ(抗体の小断片)の Tm の大規模なデータセット NanoMelt です[5]。これは既存のデータに、著者らが独自に測定したデータを追加したデータベース(640 データ)で、タンパク質の濃度、pH やバッファーなどの実験条件が揃えられています。このデータセットに対し、事例 1.と同じ物理モデルで各酵素に対して digyzme score を計算し、実験値との相関を確認します。
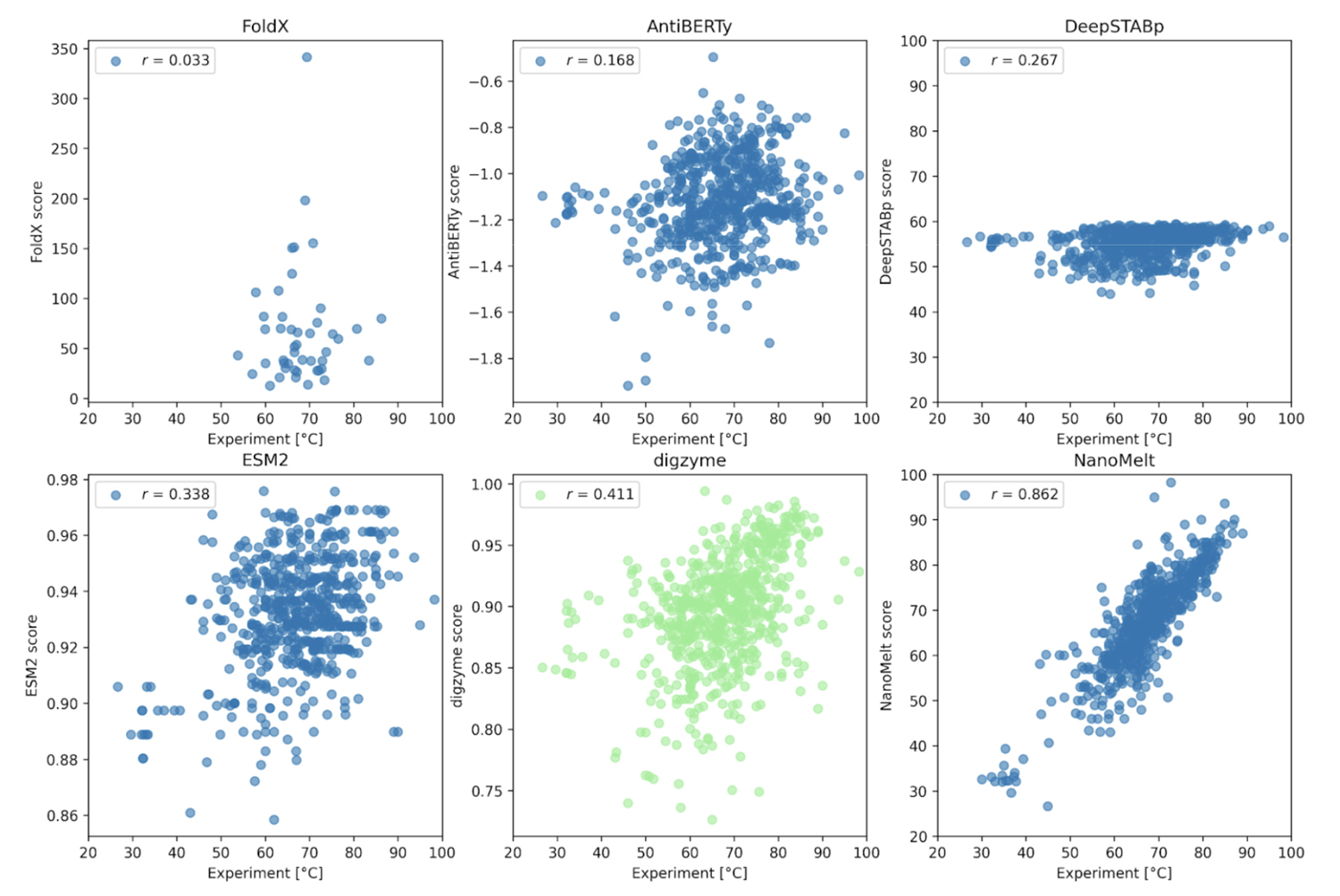
Figure 2. に示すように、 NanoMelt データセットそのものを学習対象とした結果(Figure 2下段右)以外は、実験値と予測結果の相関は存在しないか低いです。まず、事例 1.では高い相関係数を達成した物理モデルである FoldX は、ここでは相関のある予測結果を出力することはできませんでした。これに対し、digzyme では弱いながらも相関のある結果を予測することに成功しました(r = 0.411, Figure 2下段中央)。データを FoldX の予測に使用した 46 個のアミノ酸配列に限定した場合でも弱い相関が存在した(r = 0.273)ことから、これは既存の物理モデルからの適用可能範囲の拡大という意味において顕著な進歩であると言えます。AntiBERTy と ESM-2 はタンパク質の言語モデルであり、Figure 2 のスコアの実態は、アミノ酸配列の (pseudo) log-likelihood です[5]。これは配列の確らしさの指標であり、熱安定性とのある程度の相関が期待されましたが、実際には弱い相関があるのみでした(相関係数はそれぞれ 0.168 と 0.338)。従って、著者らが示すように、これらの言語モデルを使って熱安定性予測を行うとしたら、更なる学習によって専用タスクに特化したモデルを構築した方がよいでしょう[5]。一方で、Tm を予測するための専用回帰モデルである DeepSTABp の相関の低さ(r = 0.267, Figure 2上段右)についてはよく考慮する必要があります。元の論文[6]によると、DeepSTABp のテストデータに対するピアソン相関係数は 0.90 と、顕著な結果を出しています。それにも関わらず、NanoMelt データセットに対する相関係数が低かったことは、このモデルが過学習に陥っており、汎化能力に限界がある(「馴染みのないデータ」に対して無力である)ことを示唆しています。これに対し、digzyme が採用している物理モデルは物理法則という一般的な原理がモデル構築の基礎であるため、適用できるアミノ酸配列の範囲に基本的に限界はありません。実際に、DeepSTABp よりも高い相関を与えていることからも、物理モデルの優位性がある程度示されたと言えるでしょう。
さて、強い相関(r = 0.862, Figure 2下段右)を示している NanoMelt のモデルですが、過学習の有無が気になるところです。残念ながら、NanoMelt はナノボディ専用モデルであるため、任意のタンパク質配列を含む他のデータセットでその性能を再評価することは困難です。そのため、著者らはラクダのナノボディ配列のデータベースから配列を新たに 6 つ選定し、実験で Tm を測定することでモデルの再評価を行っています。配列の選定基準は以下の 3 つです:
1.NanoMelt データセットの中の最も類似している配列との不一致度が、少なくとも 30% である
2.AbNatiV VHH-nativeness スコア(配列の確からしさの指標)が 0.85 以上である
3.NanoMelt で予測した Tm が低い(Tm < 61 ℃)、もしくは高い(Tm > 73 ℃)
これらの 6 配列のうち、 Tm が低いと予測された 3 配列は発現しませんでしたが、一方で、Tm が高いと予測された 3 配列は発現し、 Tm の測定に成功しました。その予測結果との誤差は 1 ℃ 程度であり、NanoMelt の高い予測性能が示されました[5]。この結果を以って過学習の有無を断ずることはできませんが、NanoMelt モデルの適用可能範囲を把握するための手がかりにはなるでしょう。
終わりに
酵素探索や改変の文脈では、酵素の熱安定性を計算で評価し精確なランキングを作成することが、後の実験の工程を減らすために重要となります。digzyme では、汎用的な物理モデルを適用することで、この課題に挑戦しています。本ブログでは、2 つのデータセットに対する予測スコアの計算によって、開発したモデルの評価を行いました。事例 1. に示すように、変異体の熱安定性予測では実用レベルの精度を持つことが明らかになりました。一方で、任意のアミノ酸配列間の比較に関しては、事例 2. に示すように、既存の物理モデルや機械学習モデルに対する優位性が示されたものの、実験値との相関は弱く、未だ改善の余地があることもわかりました。
昨今では、 AI による酵素設計の成功例が増えてきており、生命進化の過程で取りこぼされた新規なフォールド(構造)および機能の実現が期待されていますが、その新規性が機械学習による(熱安定性などの)物性予測を困難たらしめることは想像に難くありません。そのため、本ブログで紹介したような、学習データに依存しない汎用的な物理モデルの重要性がますます高まると考えられます。digzyme では、より精確で信頼性の高いモデルを構築するために、今後も積極的に研究開発を進めていく予定です。
参考文献
[1] CAGI5 Frataxin [Link]
[2] Savojardo et al. Hum. Mutat., 2019, 40(9), 1392 [Link]
[3] Lindorff-Larsen et al. Science, 2011, 334, 517 [Link]
[4] Berliner et al. PLoS ONE, 2014, 9(9), e107353 [Link]
[5] Ramon et al. bioRxiv, 2024 [Link]
[6] Jung et al. Int. J. Mol. Sci., 2023, 24(8), 7444 [Link]
糖鎖データベースからの目的単糖を含む多糖の検索と可視化
はじめに
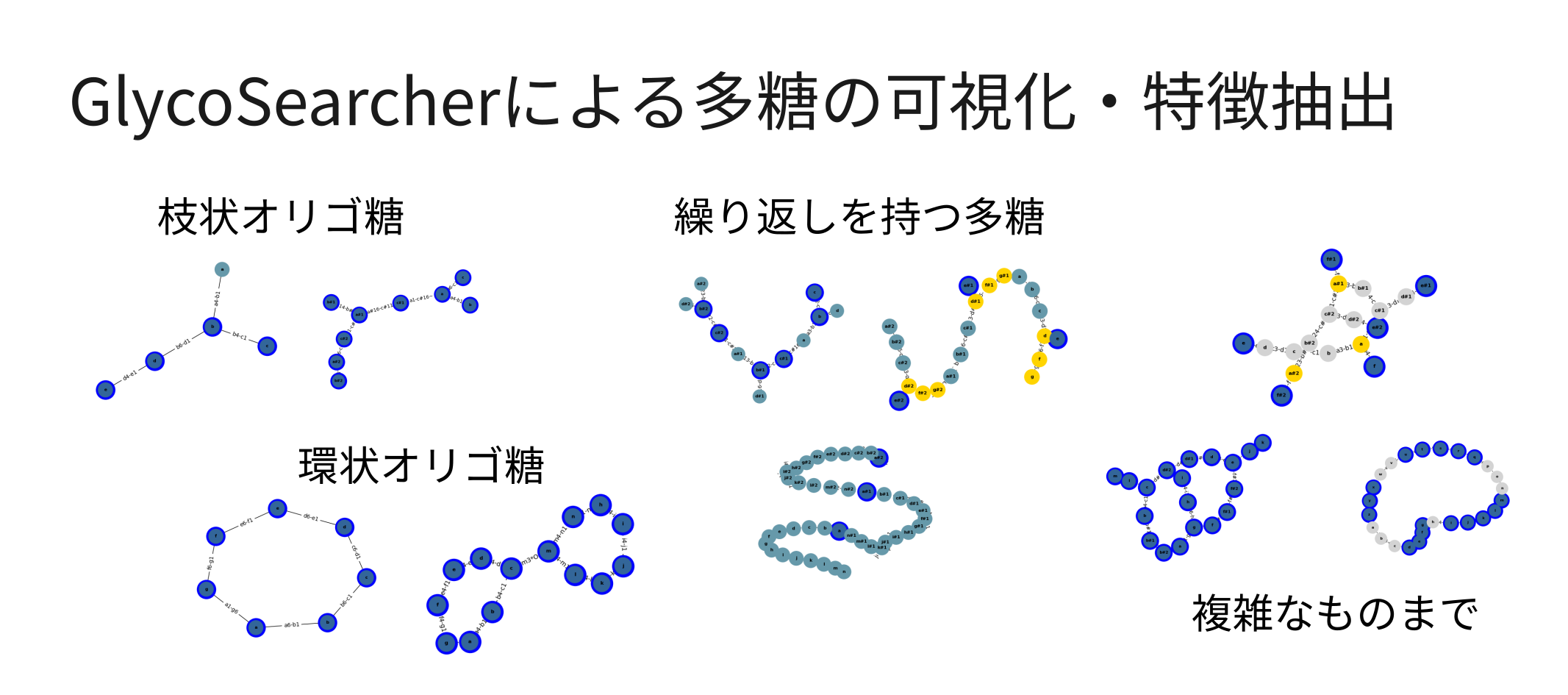
digzymeにてインターン勤務させていただいております、東京大学修士2年の高栁龍です。大学では、タンパク質のリン酸化やタンパク質立体構造に関連した研究などを行っています。今回のtechblogでは、研究開発業務の一環として新たに開発しました、目的単糖を含む多糖の網羅的検索と可視化ツールであるGlycoSearcherを紹介いたします。
近年、デンプンや食物繊維で代表されるように、糖鎖など多糖の研究と産業利用が活発となってきています。新規の糖質を開発する需要が上がってきており、その中で多糖は構造の多様性が非常に高いものとして注目されています。そこで、様々な多糖を網羅的に検索するためのツールとして、新たにGlycoSearcherを開発いたしました。
多糖の記述方式とデータベース
今回対象としている糖化合物は、既に数十万を超える数が報告されており、データベース化されています。こうした多くの糖化合物の中から目的に沿った多糖を厳選し、合成経路の探索や酵素開発などの応用につなげるには、計算処理しやすい記述方式と網羅性の高いデータベースが必要となります。
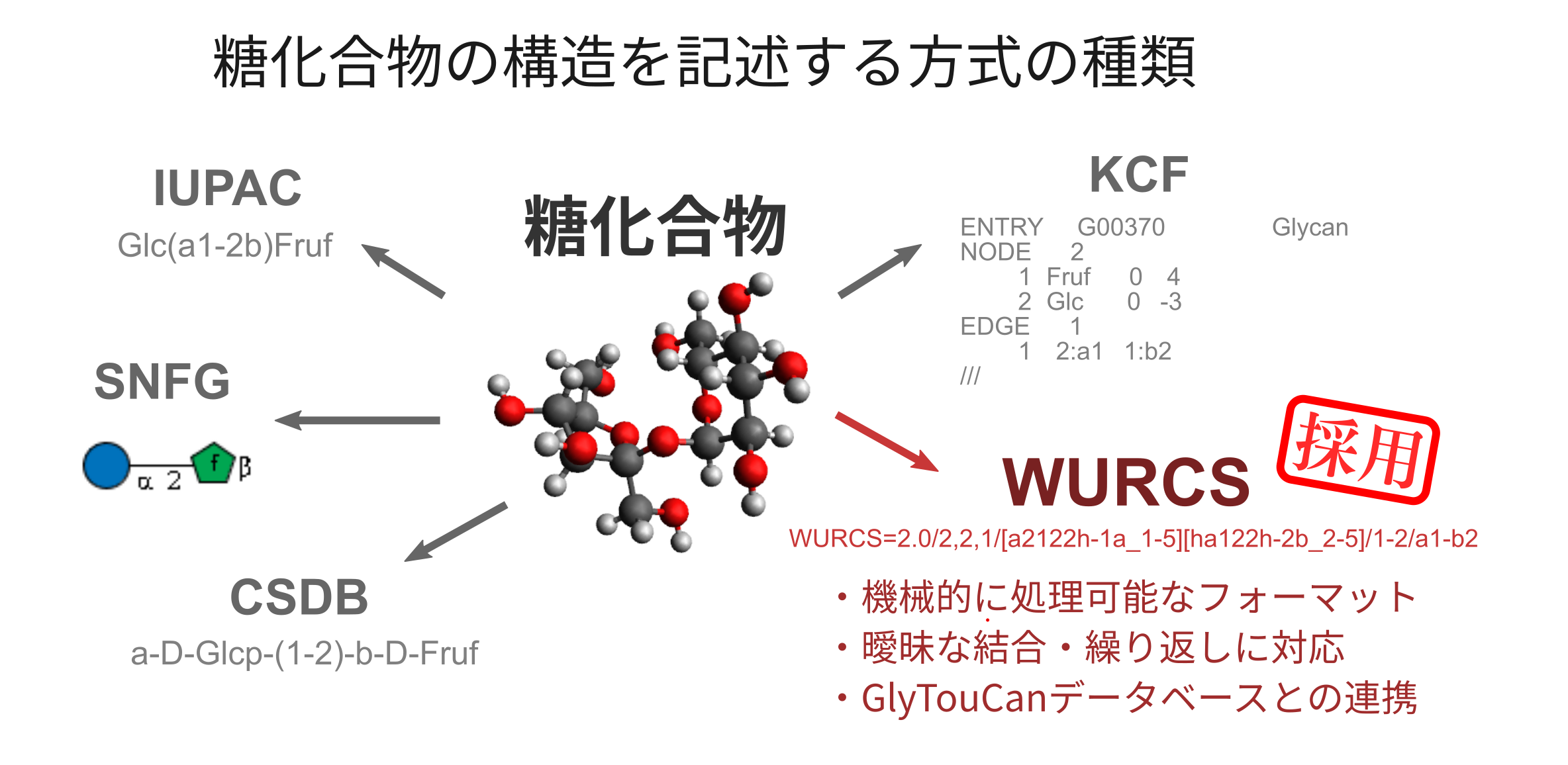
糖化合物の構造を記述する方式には様々なものが知られています(図1)。SNFGやKCFといった形式は可視化にすぐれていますが、構造情報の抽出や比較など応用的な計算処理には不向きです。一方、IUPACは、人と機械の両方が読み取り可能で簡潔な構造表現ですが、繰り返しなど複雑で曖昧な表現に対応するのは難しいです[1]。そこで、GlycoSearcherでは、計算処理に適し、繰り返し表現可能なWURCS形式、およびWURCS形式での糖化合物情報を収集したGlyTouCanデータベース[2]を採用しました。
(図1)
GlycoSearcherを用いた検索
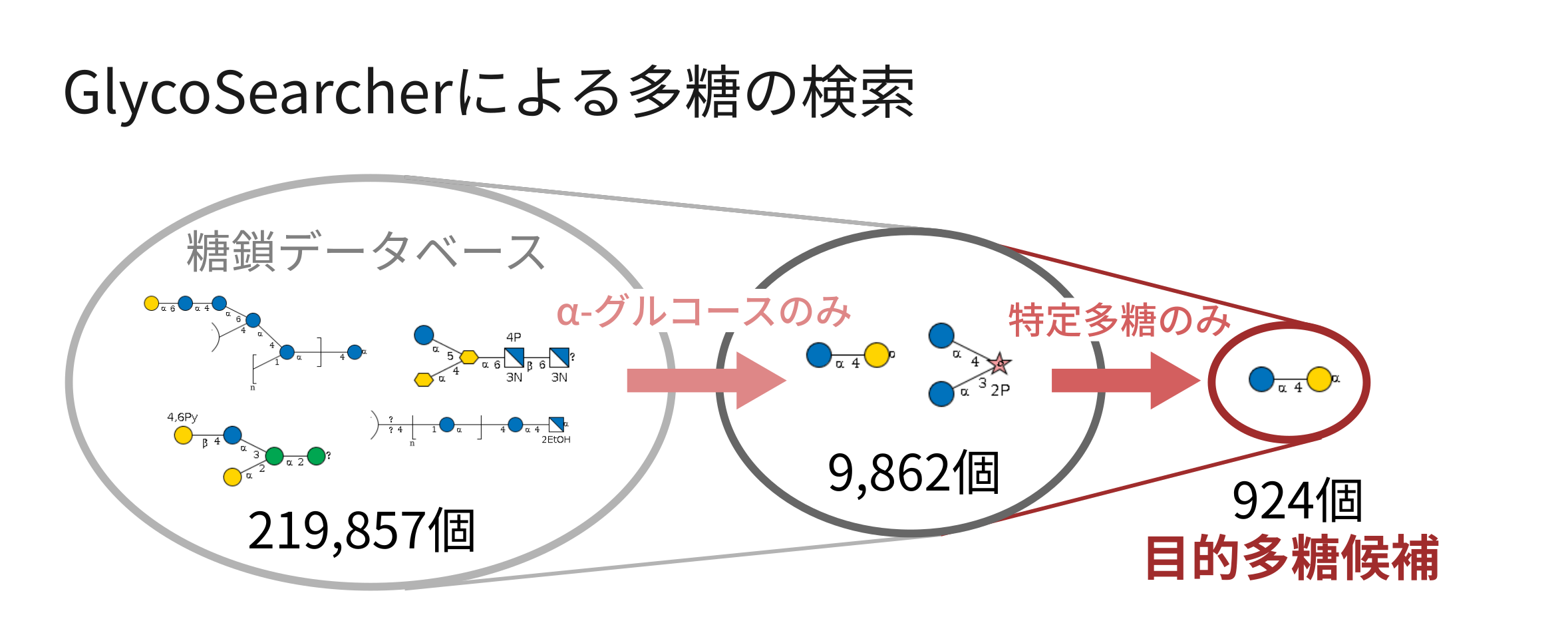
ここにGlycoSearcherでは、膨大な量の候補から目的に沿った多糖を抽出することが可能です。例えば、グルコースやガラクトースなど、特定の単糖単位を含む多糖を検索することができます。さらに、多糖を構成する単糖単位を一部のものに限定する、フィルタリング機能も兼ね備えています。これにより、ある特定の単糖を原料とし、その他特定の糖のみを用いて合成可能な多糖を列挙することができます。
以下に、α-グルコースを例に実行した結果を紹介します(図2)。219,857個ある糖鎖の中から、α-グルコースを含む多糖を検索したところ、9,862個が検出されました。さらに、構成単糖をグルコース・ガラクトース・フルクトースのみに絞ったところ、924個まで候補が減少しました。
(図2)
多糖構造の可視化と特徴抽出
得られた検索結果は、効果的に可視化され、続く応用処理へと活かすことができます(図3)。WURCS形式の多糖をグラフとして再構成することで、数千個の検索結果を数分のうちに描きだすなど、高速な可視化が可能です。また、曖昧な繰り返し回数を持つ構造についても、特定の回数だけ繰り返しを展開することで、実質的な構造を可視化するのみならず、曖昧なままでは難しい構造間の比較など、更なる計算処理へとスムーズに移行させることができます。
検索結果の多糖がグラフ化されていることから、多糖構造に対する特徴抽出も可能です。例えば、得られた多糖構造の末端にグルコース単位が存在しているか、あるいは特定の構造(モチーフ)を含むか、といった計算を行うことができます。さらに、検索によってヒットした多糖について、PubChem[3]などの各種データベースと統合させることで、その一般名や関連する酵素情報と統合させることができ、多糖を含む反応についての情報を得ることが可能です。
(図3)
終わりに
今回開発したGlycoSearcherでは、目的の多糖をデータベースから網羅的に検索し、更なる計算処理へと応用させることができました。さらに、得られた目的多糖候補から情報を抽出しその合成に関与すると予測される酵素情報を取得することで、その後の酵素デザインのワークフローにつなげる体制を整えられました。
謝辞
糖化合物に関する知識の習得を始めとしたGlycoSearcherの開発には、事業開発部の礒崎さんに大変お世話になりました。この場を借りて感謝申し上げます。
参考文献
[1] 細田 正恵, 木下 聖子.「糖鎖関連インフォマティクスへの入り口」 JSBi Bioinformatics Review, 2(1), 87-95 (2021).
[2] https://glytoucan.org/
[3] https://pubchem.ncbi.nlm.nih.gov/
ifia JAPAN 2024会場で頂戴したご質問への回答まとめ
食品事業部の村瀬です。
弊社は、2024.5.22(水)~24(金)、東京ビッグサイトにて開催された
「ifia JAPAN 2024 第29回 国際食品素材/添加物展・会議」
(食品化学新聞社主催)に出展いたしました。
展示ブースにお越しいただいた皆様、誠にありがとうございました。
この記事では、展示期間中、皆様から頂戴したご質問の中から
特に多くいただいた内容をご紹介し、回答いたします。
ぜひ最後までご覧ください。
Q:何ができる会社ですか?
A:お客様のご要望に応じて、新規酵素探索、酵素改変を行います。
従来の手法とは異なる、独自のバイオインフォマティクス技術を用いた
スピーディーな酵素開発によって、酵素メーカー、食品メーカー両者にとっての
イノベーションアクセラレーターになることができると考えています。
Q:具体事例はありますか?
A:ケミカル用途では、ユーザーが必要とする新規酵素の探索や、
酵素の大幅な活性向上に成功した事例がございます。
食品用途では、現在複数のお客様より具体的なテーマをいただき
実際にお取り組みさせていただいている状況です。

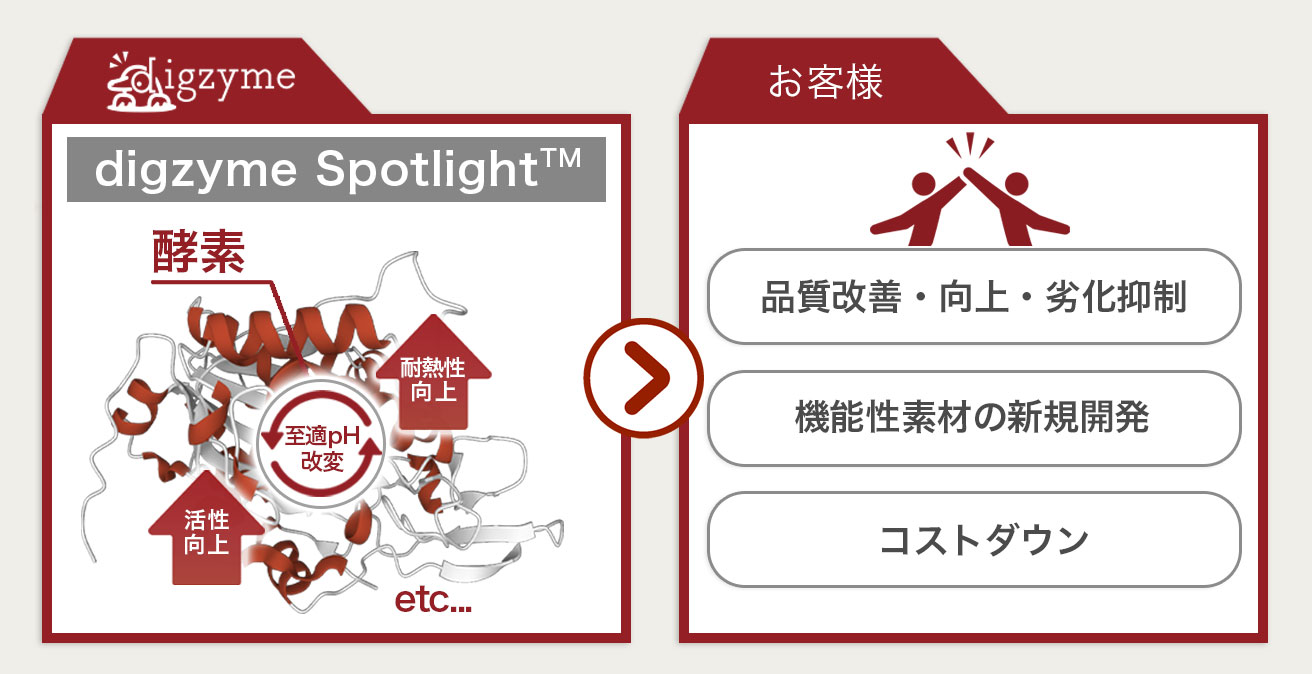
Q:digzyme Spotlight(酵素改変型プログラム)では、酵素の何の性質が改変できる?
A:活性向上、耐熱性向上、至適pHの改変などが考えられます。
基質特異性の改変は状況に応じて、digzyme Moonlight(酵素探索型プログラム)を使用します。
Q:どのような流れで開発を進める?
A:お客様の状況に応じて、スタートとゴールを設定させていただきますが
主な流れは以下となります。
1.開発コンサルティング:お客様の課題をヒアリングし、ターゲットとする酵素を選定します。
2.酵素デザイン:スーパーコンピューターを用いて、ターゲットとなる酵素のデザインをします。
3.酵素ライブラリ提供:コンピューター上でデザインした酵素を
実際に微生物を用いてラボスケールで製造し、目的に沿った酵素かを検証・確認します。
4.酵素の生産提供:製造スケールをラボからプラントへスケールアップし、
製品として酵素を安定供給できる体制を整備します。
当記事でのQ &Aは以上です。
それでは、最後までご覧いただきありがとうございました。
その他のご質問などございましたら、下記のコンタクトフォームよりお問い合わせくださいませ。
【▼コンタクトフォーム】
https://www.digzyme.com/contact/
Spotlightによる酵素変異体の活性予測精度を先行研究と比較
はじめに
事業開発部の礒崎です。弊社では酵素の活性や耐熱性などのプロパティを向上させる変異体を機械学習モデルを用いて提案するSpotlightというサービスを提供しています。様々な酵素を使って学習済みのモデルに、社内または社外から依頼を受けた目的の配列をインプットすることで活性などが向上する変異体を予測します。今回のtechblogではこのSpotlightの変異体の活性予測精度が先行研究と比較してどの程度なのか検証しました。
比較対象に使用した先行研究
Li et al., 2022では、酵素のアミノ酸配列と化合物を入力情報としてkcatを予測する機械学習モデルを構築していました。今回の比較ではこの機械学習モデルアルゴリズム(DLKcat)を用い、かつ、比較を平等にするためにSpotlightと同じ教師データであるBRENDAのkcat エントリーを使ってモデルを再構築しました。この再構築したDLKcatにより予測した変異体のkcatの値とSpotlightで予測したkcatの値のいずれが実測値とより近いか比較しました。今回使用したBRENDAのエントリーにはwild type (WT) と単変異体のみが含まれるように抽出し、変異1つに対する感度が2つのモデルでどれくらい違うかに注目して比較しました。
結果
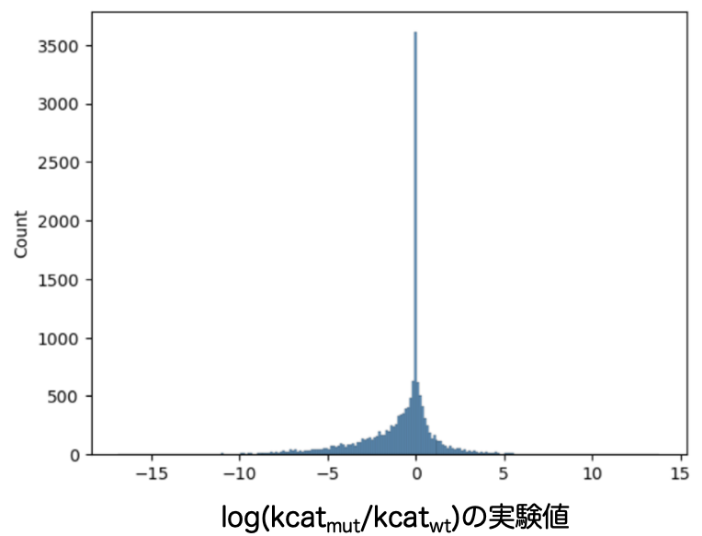
1. BRENDAのkcat (=Turnover Number)のデータを用いた学習モデルの構築
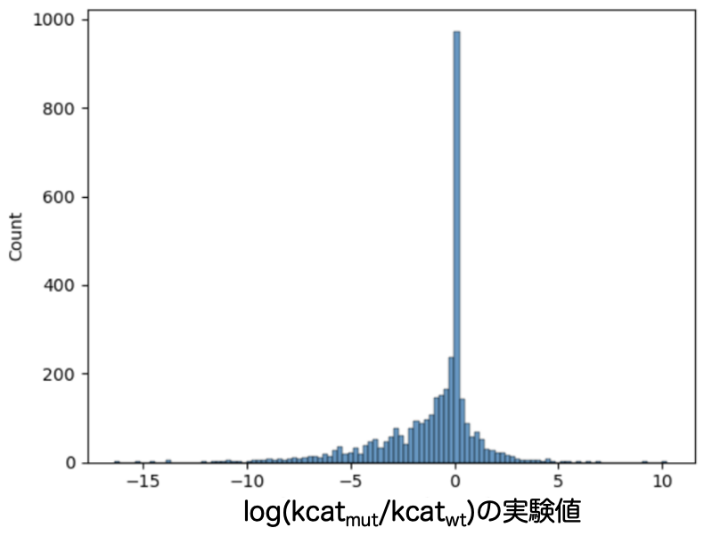
BRENDAのkcatが記載されている変異体、そのWTの配列のエントリーおよびkcatを測定した化合物の情報を抽出し、これらを酵素ファミリーに偏りが生じないように、かつ、およそ教師データ:テストデータ= 3 : 1になるように分割しました。分割後の教師データではkcatが向上しているエントリーが3969、変化しないエントリーが2985、減少しているエントリーが8296でした(図1)。分割後のテストデータではkcatが向上しているエントリーが792、変化しないエントリーが748、減少しているエントリーが1926でした(図2)。
2. DLKcat・Spotlightで予測したkcatの変異体/WT比率の評価
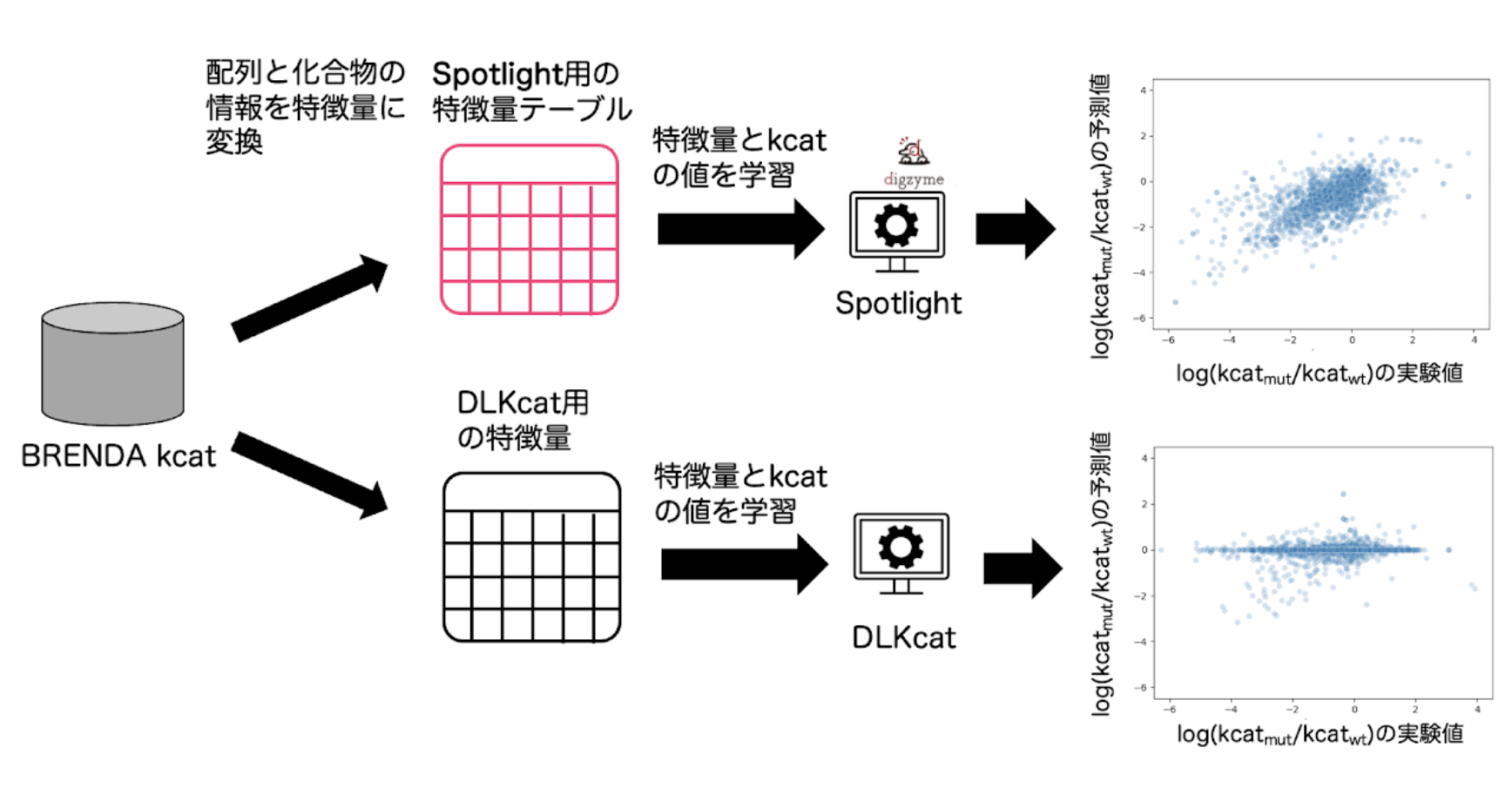
抽出したBRENDAのエントリーの情報をDLKcatが要求する特徴量の形に変換し、教師データの中のkcatの実験値と合わせて学習モデルを構築しました。Spotlightでも同様にこれらのエントリーをSpotlightが要求する特徴量の形に変換して、kcatの実験値と合わせて学習モデルを構築しました(図3)。
DLKcatで予測した変異体のkcatとWTのkcatの比率は実測値と予測値の間でピアソン相関係数が0.18でした(図3)。DLKcatにおいて予測した変異体とWTのkcatの比率が実測値と良く相関しなかった理由は、DLKcatでは特徴量として配列の全長をベクトルに変換しているため1アミノ酸の違いが特徴量に現れづらくなっているからであると考えています。Spotlightで予測した変異体のkcatとWTのkcatの比率は実測値と予測値の間でピアソン相関係数が0.66でした(図3)。弊社のSpotlightでは特徴量に変異体としての性質を大きく反映できる工夫をしてあるため、単変異体のエントリーであってもWTからの1変異による変化を正確に予測することができています。
終わりに
弊社のSpotlight™では先行研究と比べて、単変異体というWTから1アミノ酸しか違わないようなケースでも、その変化を正確に反映してより実験値に近い値を予測可能であるということが明らかになりました。
謝辞
今回の酵素活性予測の精度比較には以下の論文のデータを利用させていただきました。
Li et al., (2022) Deep learning-based kcat prediction enables improved enzyme-constrained model reconstruction. Nature Catalysis.
